
2022-07-25


《biology》
影响因子:5.168
近日,派森诺生物与四川省农业科学院合作,在生物学领域的《biology》发表新研究成果!本研究对福建和江西的采集的两只Haplodiplatys aotouensis的线粒体基因组进行了测序,结果表明该属的线粒体基因组存在种内变异和广泛的基因重排事件。本研究的发现可为H.aotouensis分子多样性和线粒体进化提供新的信息。
研究背景
革翅目,俗称蠼螋,是陆地昆虫中一个相对较小的原始类群,在世界范围内现存1900多种。蠼螋的尾羽呈钳状,无节,蠼螋成虫期的特征很容易与其他昆虫区分开Haplodiplatys aotouensis Ma & Chen, 1991,原产于中国东南部福建省,后被收录在Chen & Ma专著(2004)。此后,H. aotouensis的相关研究未见开展。尽管在形态学和分子数据上做了大量的研究,但关于革翅目的系统发育位置以及它们之间的内在联系仍未明确。与传统的形态学研究相比,对蠼螋遗传性状的研究相对薄弱。与其他昆虫相比,革翅目广泛使用的遗传标记线粒体基因组的研究甚少。迄今为止,只有6个物种的完整或接近完整的线粒体基因组被测序和分析。在Chen(2022)中总结了蠼螋的一些共同的染色体组特征和系统发育的初步结果,但需要更多的染色体组数据进一步证实。本研究收集了来自两个不同地理位置的样本,对H. aotouensis线粒体基因组进行了测序和分析。本研究旨在探讨蠼螋的线粒体基因组变异、常见的线粒体基因组结构特征及系统发育关系。
研究材料与方法
1、实验材料
2022年3月福建武义山和2021年5月江西武义山分别采集了两个样品,经鉴定这两个物种为H. aotouensis。
2、分析内容
线粒体基因组测序+组装+注释、密码子偏好性分析、Ka/Ks分析、重复序列分析、进化树构建。
研究结果
线粒体基因组结构和核苷酸组成
两个地理区域获得的两个线粒体基因组高度相同(99.4%),这可以证实它们是同一物种。福建的H. aotouensis的线粒体基因组长度为16134 bp,而江西的线粒体基因组长度为16222 bp。两个线粒体基因组都由标准的37个基因组成(13个PCGs, 22个tRNA,2个rRNA基因),但它们在以下几个方面存在差异:cox1、cox2和nad6基因分别在951、300和384bp上有一个不同的碱基;江西样品的rrnS基因在中段比福建样品多一个碱基;多数的J链有24个基因,少数的N链有剩下的14个基因。在该属的线粒体基因组中,共发现4个重叠区域,大小在1 - 7 bp之间。在atp8和atp6之间有最长的重叠区域。还有23个基因间隔区,大小从2到178 bp不等,最长的基因间隔区位于trnS2和nad1之间。两个H.aotouensis线粒体基因组呈现高度AT偏向性,福建样品和江西样品的AT含量分别为71.7%和71.9%,37个线粒体基因中的每个基因都具有丰富的AT含量,在trnK中为63.6%,在trnC中为86.6%。在两个线粒体基因组中,AT skew为负(-0.1),而GC skew为正(0.3)。
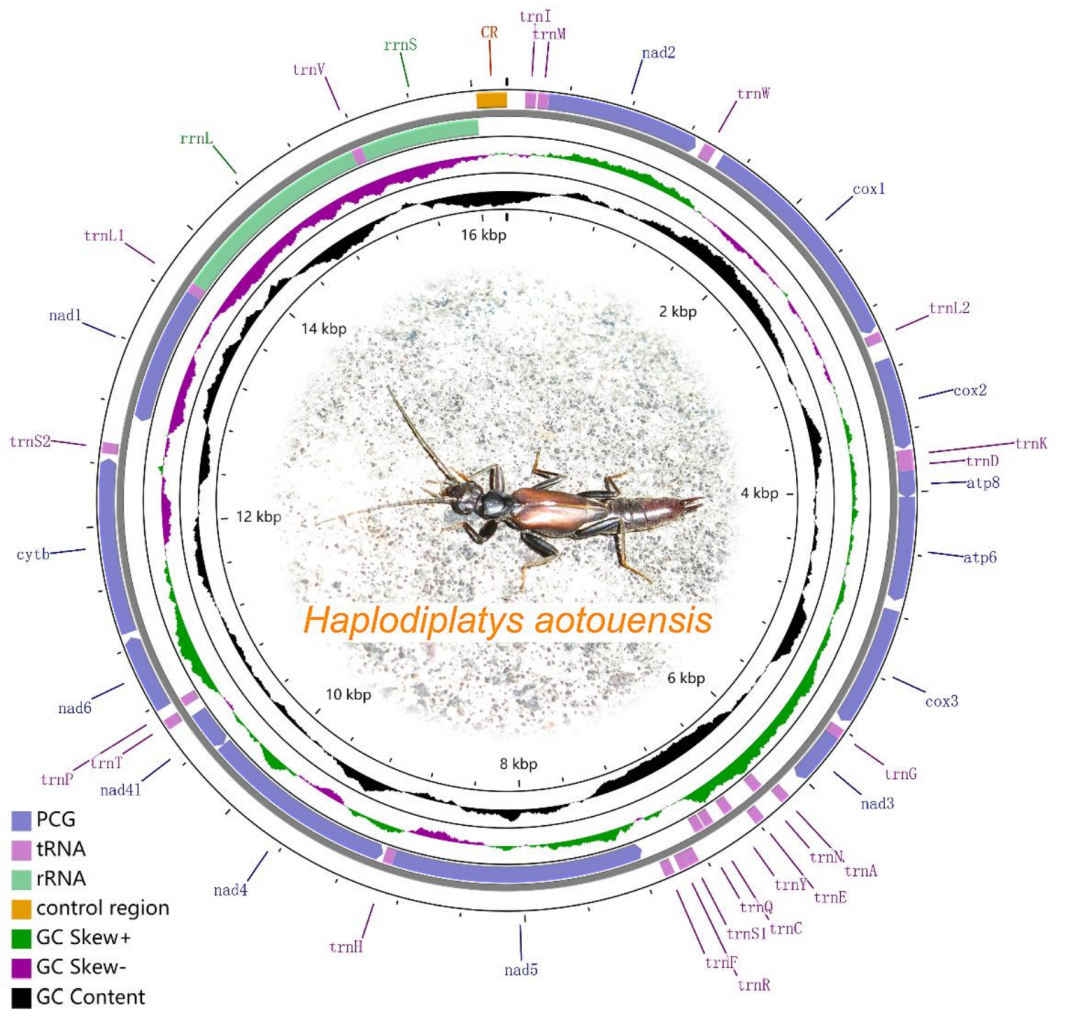
图1 线粒体基因组圈图
基因重排
两个H. aotouensis线粒体基因组中的13个基因顺序与所有已测序的蠼螋和D. yakuba的祖先节肢动物线粒体基因排列顺序相同且基因结构保守。而trnI-Q-M、trnW-C-Y和tRNA -R - N - S1 - E - F三个基因簇中的tRNA基因均发生重排,且在已测序的蠼螋中均未发现这种重排模式。CREx分析表明,H.aotouensis的线粒体基因组的基因顺序与D.yakuba的线粒体基因组的祖先类型不同,按照如下步骤进行重排的:trnC和trnY的初始转位;trnF和trnN的后续逆转; 以及trnY-trnC、trnR、trnQ和trnS1的两个最终串联复制和随机损失事件。
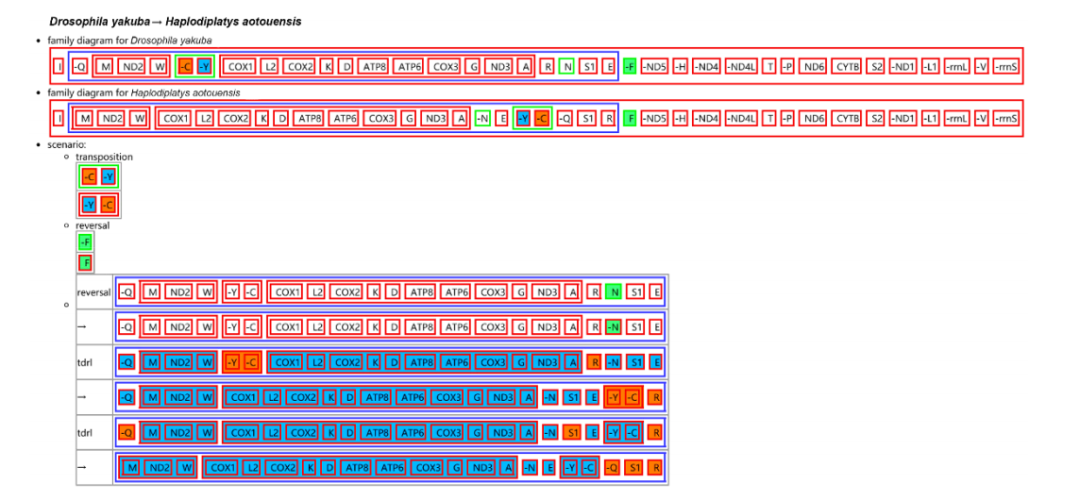
图2 H.aotouensis进化过程中线粒体基因重排
编码基因
所有PCGs都以标准的ATN起始密码子(ATT、ATC和ATG)开始,以完全终止密码子TAN (TAA或TAG)结束。RSCU结果显示在氨基酸编码密码子中使用频率最高的是TTA (Leu)、TCT (Ser)和CCT (Pro)。Ka/Ks结果表明,nad4l的进化速率最高,其次是cox1、nad4和nad6。不同地理样品中cox1、cox2和nad6的Ka/Ks比值存在差异。计算结果还显示,福建样品中cox1和cox2的进化速率略高,江西样品中nad6的进化速率略高。4个基因的比值大于1,说明它们是在正向选择下进化的。其余9个PCGs的Ka/Ks比都低于1,表明这些PCGs中存在纯化选择。
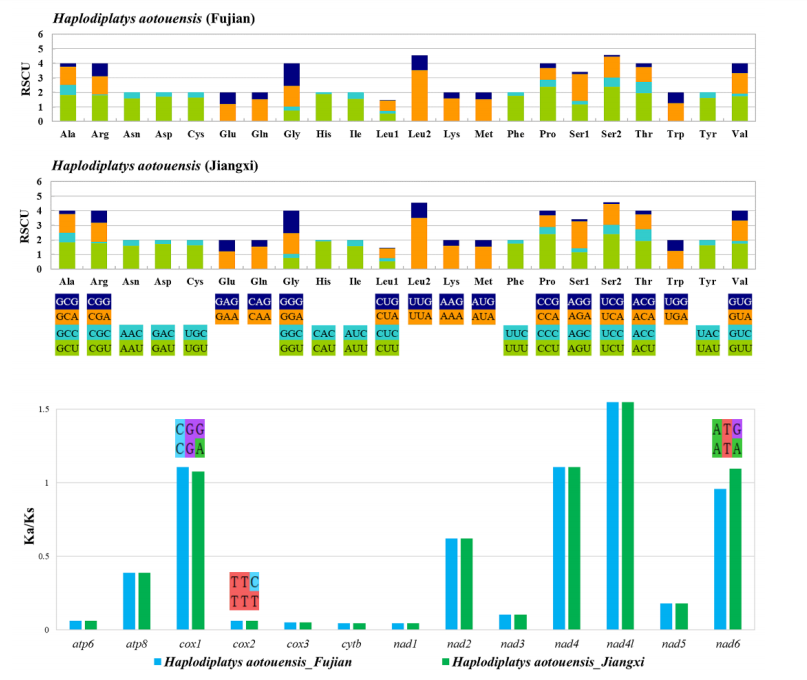
图3 不同地区的H. aotouensis线粒体基因组的RSCU和Ka/Ks分析
tRNAs, rRNAs and the Control Region
两个H. aotouensis的线粒体基因组都包含22个典型的tRNA基因,这些基因的核苷酸序列是两两相同的。这些tRNA的大小从66 - 79 bp不等,最长的tRNA基因为trnW。tRNA基因的反密码子与其他已测序的蠼螋相同。大多数tRNA基因预测的二级结构都是典型的三叶草结构,而trnS1的二氢尿嘧啶臂被缩减为一个小环。在16个tRNA基因的二级结构中共发现37个不匹配的碱基对,它们都是G-U对不匹配。大核糖体RNA(rrnL)基因和小核糖体RNA(rrnS)基因位于trnL1与控制区之间的保守位置。福建样品和江西样品的rrnL基因长度为1408 bp, AT含量均为75.4%。两种有丝分裂基因组的rrnS基因长度略有差异,福建样品为814 bp,AT含量为75.4%;江西样品为815 b, AT含量75.5%。
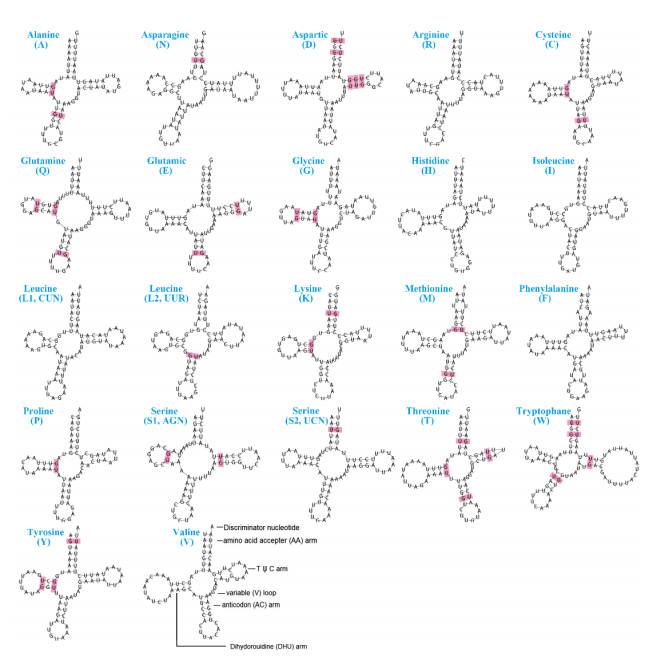
图4 tRNA的二级结构
两个样本的控制区均小于200 bp,福建样品控制区长139 bp, AT含量为77%;江西样品控制区长197 bp,AT含量较高,达83.8%。两个对照区域的核苷酸序列在1-93和94-139位置相同。在两个对照区域的50端附近检测到3个串联重复序列,每个重复序列包含8个核苷酸,即TACGCGTA。在两个对照区域的30端附近也发现了一个poly-[TA]n延伸,福建的样本长8 bp (4 TA单位),江西的样本长66 bp (33 TA单位),两个控制区之间的长度差异是由于TA单元数量不同造成的。
进化树分析
为了获得更可靠的系统发育结果,本研究排除了D. flavicollis中缺失的cox1、cox2和nad2。从饱和度图可以看出,atp8、nad4l和nad6和所有PCGs的第三个密码子,以及这两个rRNA基因已经饱和,并被排除在核苷酸数据集之外。最终的核苷酸数据集包含来自7个PCGs的4542个碱基。氨基酸数据集由2271个氨基酸组成,这些氨基酸来自7个不饱和PCGs,包括它们的第三个密码子位置。在6个系统发育树中,有5棵具有相同的拓扑结构。在所有树图中,本研究测序H. aotouensis均聚在一起。
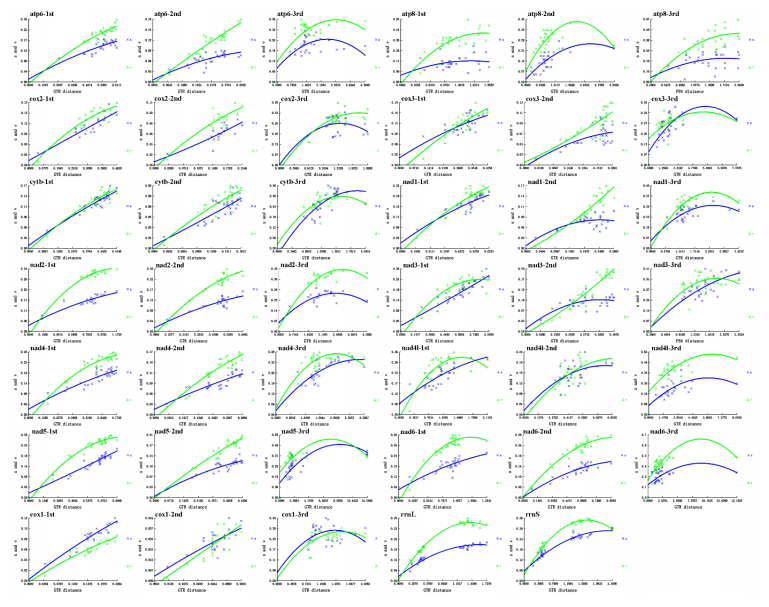
图5 PCGs的每个密码子位置和rRNA的每个基因替换饱和图
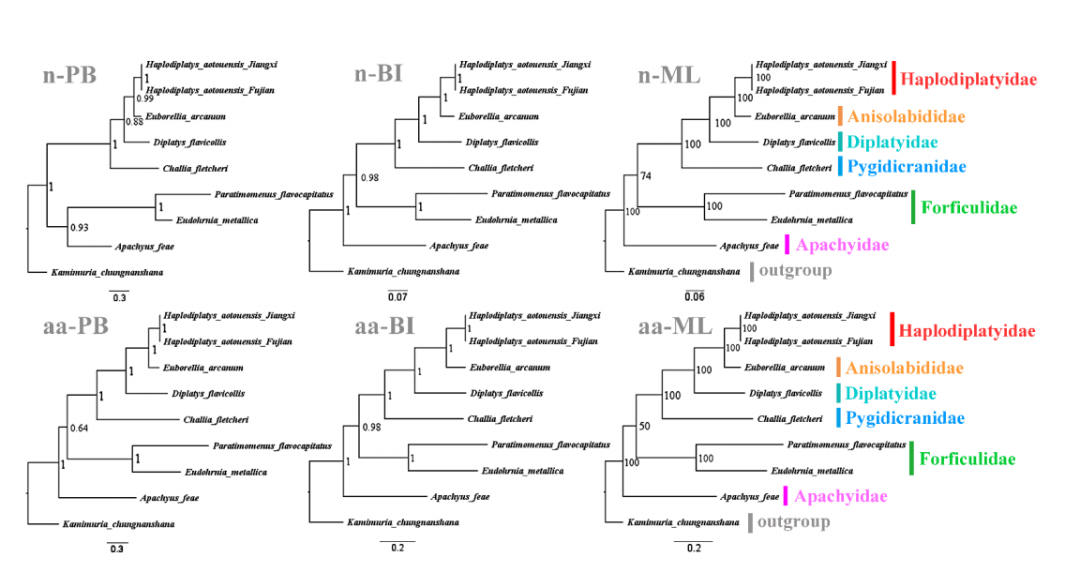
图6 进化树
结 论
本研究发现了一种中国蠼螋种内线粒体基因组变异和广泛的基因重排。然而,目前对蠼螋线粒体基因组特征的研究还很少。线粒体基因组数据有望解决革翅目系统发育问题,未来将结合核标记对革翅目线粒体基因组进行更全面的采样和测序,重建更可靠的系统发育。
本研究的denovo测序由上海派森诺生物科技有限公司完成。如需进一步讨论,欢迎发邮件或者致电我们哟(邮箱地址:microsupport@personalbio.cn,联系电话:025-56165883-832)!
文章索引:Liu H L, Chen S, Chen Q D, et al. The First Mitochondrial Genomes of the Family Haplodiplatyidae (Insecta: Dermaptera) Reveal Intraspecific Variation and Extensive Gene Rearrangement[J]. Biology, 2022, 11(6): 807.