
2022-09-06

《Journal of Hazardous Materials》
影响因子:14.224
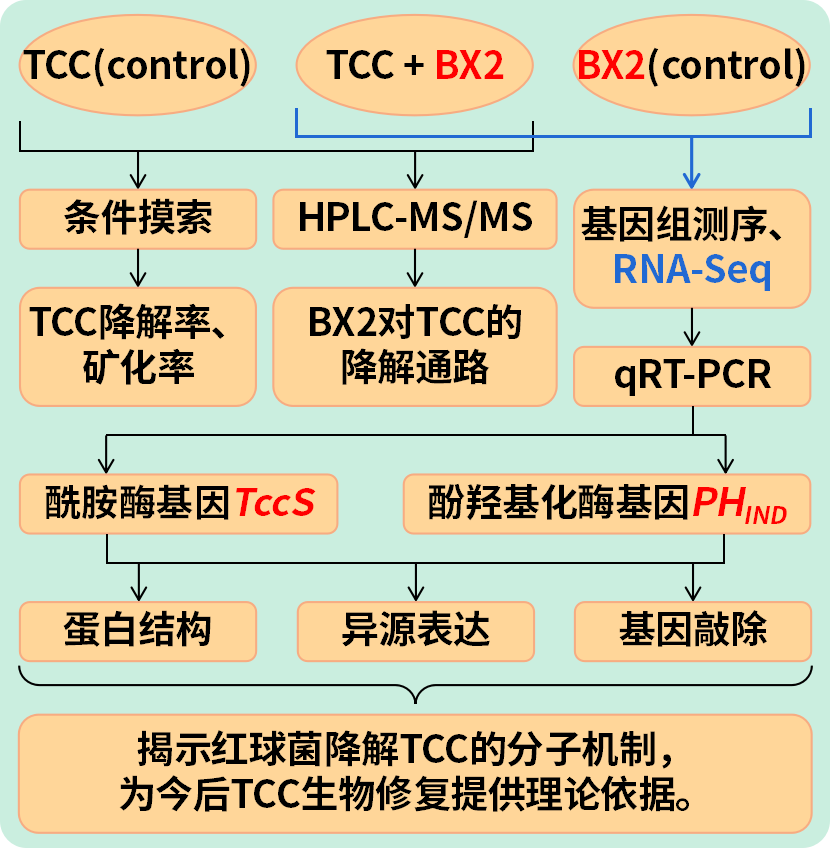
技术手段:HPLC-MS/MS,基因组测序,RNA-seq,qRT-PCR;
东北农业大学于近期在《Journal of Hazardous Materials》上发表了一种酰胺酶和一种新型酚羟化酶催化红球菌降解抗菌剂三氯卡班的研究成果。
三氯卡班(Triclocarban,TCC)是一种广谱抗菌剂,由于较高的杀菌效果而被广泛应用于药品和个人护理产品中。因为TCC具有疏水的特性以及污水处理厂去除不完全的问题,TCC是自然环境中检测最广泛的污染物之一。而且由于TCC对哺乳动物、水生生物和陆生生物有毒力,长期暴露于生态系统中的TCC及其脱氯代谢物已导致健康问题。因此,人们对TCC的污染问题越来越关注。
与物理和化学技术相比,微生物降解转化TCC具有几个优点,包括相对较高的效率和生态友好的特点。然而,考虑到TCC的顽固性和抗菌性,对有效降解TCC的微生物种类的研究有限。到目前为止,已发现的降解TCC的细菌包括鞘氨醇单胞菌YL-JM2C,苍白杆菌TCC-1、TCC-2、MC22和假单胞菌MC46,然而这些菌株都不能将TCC作为唯一的碳源,也没有表现出对TCC的耐受性增加。
尽管初步研究表明细菌对TCC有良好的生物降解迹象,但真正的生物降解通路尚未得到确凿证据的证实。因此,本研究以实验室先前分离出的以TCC为唯一碳源生长的红球菌BX2为研究对象,探究BX2菌株对TCC的降解能力和通路、推测并验证功能基因,为今后TCC生物修复提供理论依据。
研究结果
使用RSM优化培养条件并进行验证
首先,探究了温度、pH、接种量、培养时间对TCC降解率的影响,最终确定BX2菌株的最佳培养条件为30.05℃、pH为7.05、接种量为3.42%、培养时间为5.36d。在最优培养条件下,BX2菌株在5天内细胞生长迅速增加,对10mg/L TCC的降解率达76.8%,总有机碳(TOC)的去除效率为56.5%。结果表明,BX2菌株具有较强的生物降解和矿化活性,是TCC污染环境修复的重要候选菌株。
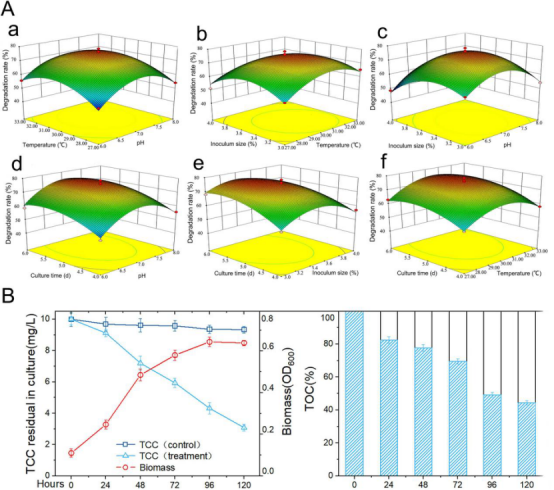
图1 A:不同因素对BX2菌株生物降解活性的交互作用的三维响应面图。B:菌株BX2的生长情况和对TCC的降解、矿化情况。
BX2菌株的TCC生物降解通路
采用HPLC-MS/MS法对BX2菌株降解TCC不同时间点的样品进行分析,结果显示,对照组只观察到母质TCC,未检测到其他代谢物;处理组中除TCC外陆续鉴定出6个主要化合物。基于HPLC和HPLC-MS/MS鉴定的代谢物以及前人研究的结果提出了BX2菌株降解TCC的通路。在此通路中,TCC降解的主要步骤是酰胺键的水解,生成产物I(DCA)和II(4-CA),产物I没有进一步降解。随后,产物II被脱胺和羟基化,生成产物III(4-chlorocatechol)。产物III通过邻位裂解进一步降解形成产物IV(3-chloro-cis-cis-muconic acid)。产物IV经过一系列氧化脱酯化反应生成产物VI(β-ketoadipic acid),然后进入三羧酸(TCA)循环。
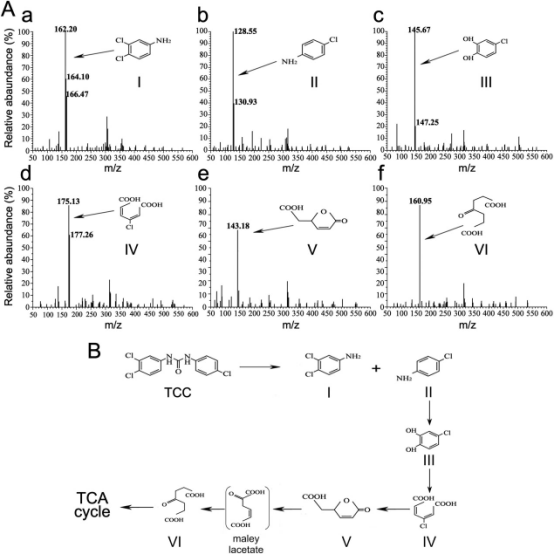
图2 A:TCC生物转化产物的HPLC-MS/MS分析质谱。B:BX2菌株对TCC的降解通路。
BX2菌株的全基因组和转录组均显示有降解TCC的功能基因
前人研究发现,酰胺酶TccA催化TCC水解。根据BX2菌株的基因组测序数据,鉴定出16个编码酰化酶的基因。根据氨基酸序列对已确认的酰胺酶基因TccA(KU753911)和这16个基因进行了系统发育树分析,其中酰胺酶基因TccA、chro-orf00841、chro-orf04781和chro-orf04940聚在一起,表明它们的功能可能类似。转录组结果显示,TCC处理导致58个基因上调,53个基因下调。细胞中上调的基因促进了代谢,但下调的基因表明TCC对BX2菌株仍有毒性,细胞代谢略有下降。与对照组相比,处理组编码苯酚羟化酶的基因chr-orf07198的表达量显著增加(log2fold-change(T/C)为4.61)。
qRT-PCR验证TCC降解相关功能基因
对上述4个候选基因进行qRT-PCR验证,结果显示,酰胺酶基因chr-orf04781和chr-orf00841在24-84h时的mRNA表达量略高于对照组,但在96h时显著低于对照组,因此这两个基因可能不参与TCC的降解;在24-96h时,处理组中chr-orf04940(命名为TccS)和chr-orf07198(命名为PHIND)基因的表达量显著增加,与预期的降解通路一致,表明TccS和PHIND在BX2菌株降解TCC中起着至关重要的作用。
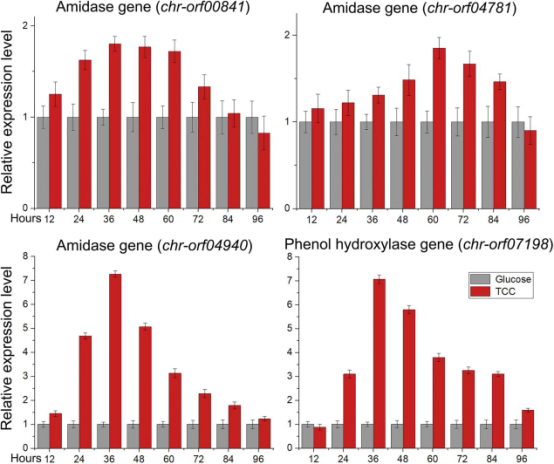
图3 BX2菌株在加TCC和不加TCC情况下培养12-96h后,基因的相对表达水平。
TccS和PHIND的活性位点和底物结合口袋
为了确定上述基因编码的TccS和PHIND的功能,本研究通过分子对接预测了相应底物与蛋白质之间的相互作用。TccS包含一个底物结合口袋。与酰胺转移酶蛋白的序列比对显示,TccS包含一个由Gly275-Gln400-Lys402组成的催化三联体。此外,4-CA可以与蛋白活性位点残基(Ser363)形成氢键相互作用。活性位点氨基酸残基形成了PHIND的底物结合口袋。TCC的氢键供体和受体基团可以与TccS的催化三联体Gly275-Gln400-Lys402形成氢键相互作用。4-CA通过氨基氢键固定在PHIND的活性位点上。从结构上看,TccS和PHIND可能分别具有催化TCC及其中间体4-CA降解的潜力。
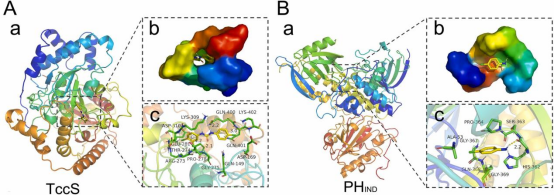
图4 A:TccS与TCC的结合方式。a:复合体的三维结构。b:活性部位的表面,TCC化合物呈现为黄色。c:复合物的详细结合模式,蛋白质骨架呈现在管中并呈现彩虹色。B: PHIND与4-CA的结合方式。黄色短划线表示氢键距离或π堆积。
TccS和PHIND的异源表达、纯化及功能分析
将TccS和PHIND基因克隆到pET28a(+)中,并在大肠杆菌BL21(DE3)中表达。TccS蛋白分子量为50.411kDa,属于酰胺酶家族。纯化后的TccS蛋白60min降解52.0%的10mg/L TCC,180min降解96.4%,240min完全降解。PHIND蛋白分子质量为38.498kDa,属于多组分单加氧酶家族。纯化后的PHIND蛋白60min降解42.3%的5mg/L 4-CA,240min降解97.2%,300min完全降解。采用HPLC-MS/MS分析纯PHIND产生的4-CA降解产物。检测到母体4-CA底物和一种子产物,子产物与产物III相对应,如我们提出的TCC降解通路所示。
根据系统发育分析和序列比对,TccS与其它经鉴定的酰胺酶基因的同源性较低(27.30-42.07%);PHIND与不同的酚羟基化酶具有较低的同源性(23.38-34.91%)(数据来自NCBI SwissProt蛋白数据库)。
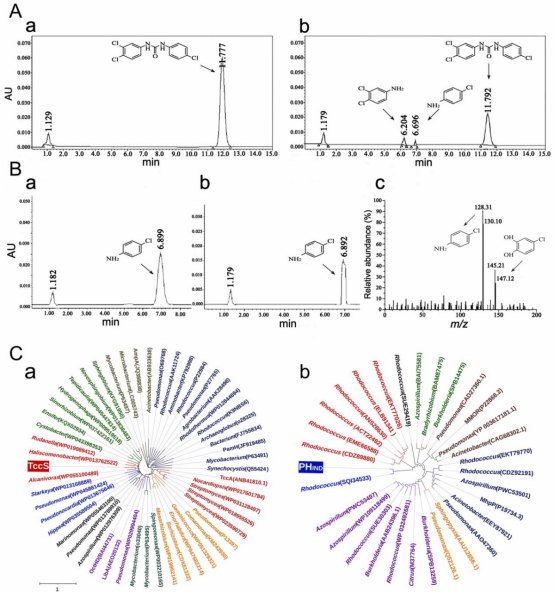
图5 A:HPLC检测TccS催化的TCC降解产物。B:HPLC检测PHIND催化的4-CA降解产物。C:基于TccS(a)和PHIND(b)的氨基酸序列及其同源蛋白构建的系统发育树。
敲除BX2菌株的TccS和PHIND基因
为了验证TccS和PHIND在BX2菌株TCC分解代谢中的生理作用,通过同源重组构建了TccS和PHIND的缺失突变体BX2ΔtccS和BX2ΔPHIND菌株。研究了野生型BX2菌株和突变株BX2ΔtccS的TCC降解速率和生长情况,以及野生型BX2菌株和突变株BX2ΔPHIND的4-CA降解速率和生长情况。结果表明,BX2菌株中TccS参与了TCC的降解,PHIND参与了4-CA的降解,且缺失TccS和PHIND不足以完全抑制TCC和4-CA的降解,这可能与其它相关基因的存在有关。综上所述,TccS和PHIND是BX2菌株TCC代谢的关键酶,即TCC通过TccS转化为DCA和4-CA,然后PHIND将4-CA羟基化为4-氯邻苯二酚(产物III)。
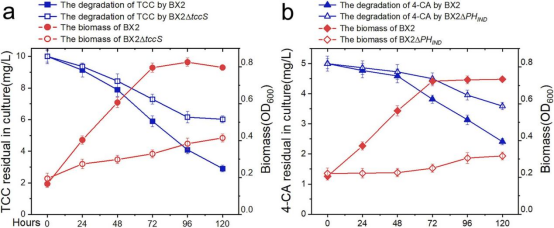
图6 BX2ΔtccS(a)菌株降解TCC,BX2ΔPHIND(b)菌株降解4-CA。
结 论
本研究发现红球菌BX2对TCC具有有效的降解和矿化能力。降解代谢产物分析表明,菌株BX2存在一条进入TCA循环的TCC生物降解通路。对BX2全基因组和转录组的挖掘发现了TCC降解相关基因。通过异源表达和基因敲除分别验证了TccS和PHIND基因分别是TCC及其中间体4-CA转化的必要基因。本研究为研究TCC的微生物降解机制提供了新的思路,为进一步利用微生物修复TCC污染提供了参考,并为今后酶资源的应用提供了理论依据。
本研究的基因组和转录组测序分析由上海派森诺生物科技有限公司完成。
参考文献:
Li C, Sun Y, Sun G, Zang H, Sun S, Zhao X, Hou N, Li D. An amidase and a novel phenol hydroxylase catalyze the degradation of the antibacterial agent triclocarban by Rhodococcus rhodochrous. J Hazard Mater. 2022 May 15;430:128444. doi: 10.1016/j.jhazmat.2022.128444.