
2022-10-17

《Metabolites》 影响因子:5.581
近日,云南大学生态与环境学院在生物学领域的《Metabolites》发表新研究成果!研究对C. pseudotenuipes进行了全基因组测序,并与三个同属物种的基因组进行了比较分析,确定其基因组的特征。此外,还探索了虫草属基因组中的次生代谢物(SMs)和生物合成基因簇(BGCs)。本研究为靶向基因组挖掘提供了可能,如基因敲除、微生物基因的导入或异源表达、启动子调控、突变诱导等,以唤醒沉默的BGC并合成更多新的生物活性SMs,进而用于新药研发。
研究背景 C. pseudotenuipes是2022年4月发布的一种新真菌,该物种是根据其形态与C. tenuipes的相似性命名的。从分类学上讲,该种与C. tenuipes及其类似类群(C. cicadae和C. militaris)有亲缘关系。虫草属具有多种生物活性成分,包括虫草素、虫草多糖、生物碱、虫草酸、喷司他丁、ophicordin、白僵菌素等,这些生物活性成分以不同方式发挥作用。 基因组挖掘是一种从基因组数据中自动检测和注释生物合成基因簇(BGC)的方法。随着基因组测序的发展,如新化合物的鉴定和表征,增加了基因组挖掘的应用范围。前人通过基因组挖掘发现,C. militaris的cns1-cns3基因簇产生虫草素和戊烯酮,其中cns1和cns2是合成虫草素所必需的,而cns3是戊烯酮合成所必需的。而且,C. militaris基因组内非核糖体肽类合成合酶(NRPS;T-C-A-T-C-C-A-T-C-A-T-C-C),多聚乙酰合酶(PKS;KS-AT-DH-MT-ER-KR-ACP),酰基转移酶,和ATP依赖长链脂肪酸酰基辅酶等催化白僵菌内脂合成,该化合物是通过在构巢曲霉中异源表达产生的。为了进一步探索C. pseudotenuipes产SM的能力,对其进行了全基因组测序和分析,并将其与虫草属其中的菌株进行了基因组比较和产SM能力比较分析。
研究材料与方法 1、实验材料 虫草属菌株分别取自越南沙巴、云南省贡山县、云南省楚雄市、云南省昆明 2、测序平台 Illumina NovaSeq 3、分析内容 真菌基因组测序+组装+注释、次生代谢产物合成基因簇分析、共线性分析、进化树构建。
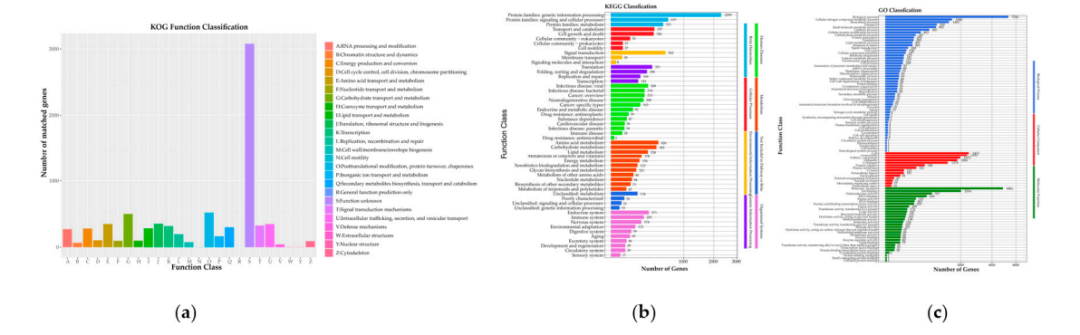
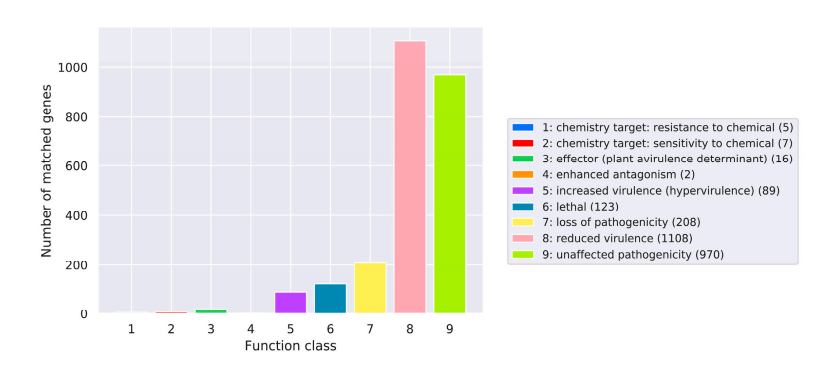
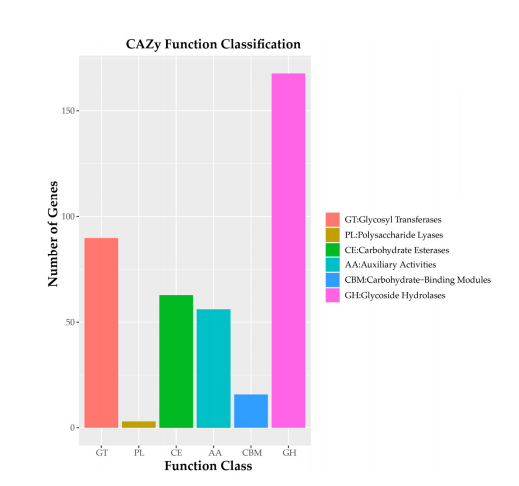
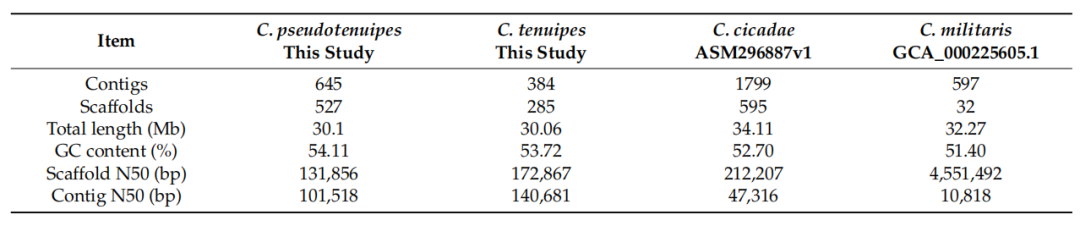
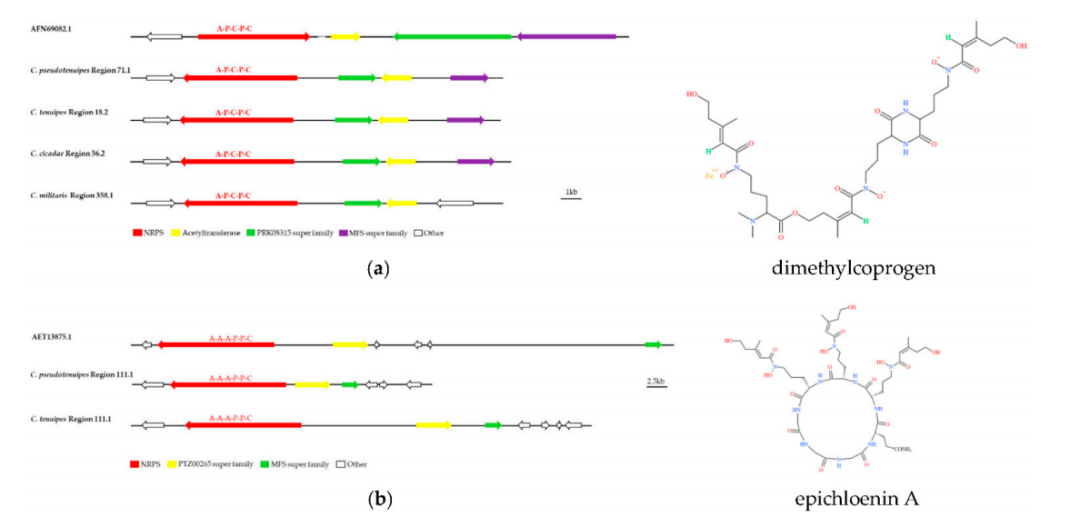
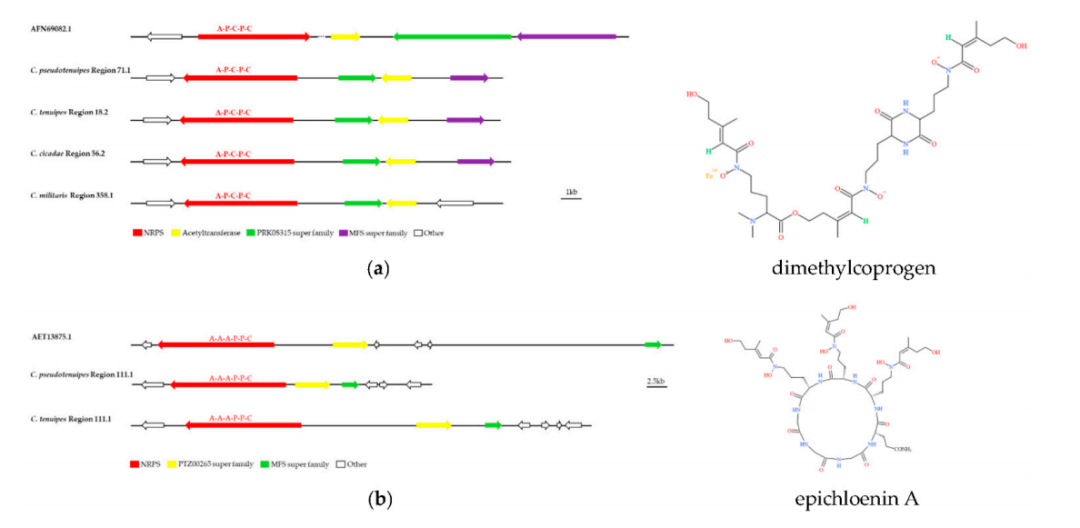
研究结果 1. C. pseudotenuipes基因组特征 C. pseudotenuipes基因组全长30.1 Mb,包含527个scaffold,N50为131,856 bp,GC含量为54.11%。基因预测得到8705个蛋白质编码基因,基因序列总长度为13.97 Mb,平均序列长度为1605.2 bp(表1)。此外,还预测出了124 个tRNA、31个rRNA和37 个snRNA等非编码RNA。 表1 C. pseudotenuipes基因组组装与功能注释结果 EggNOG数据库注释结果表明,大多数预测基因在功能上与“功能未知”(3079)、“翻译后修饰、蛋白质转换”和“碳水化合物运输和代谢”相关(图1a)。KEGG功能聚类揭示了用于遗传信息处理(2295)、信号和细胞处理(619)以及信号转导(562)的蛋白质家族(图1b)。GO注释表明,生物过程(5316)、细胞氮化合物代谢过程(1566)和生物合成过程(1425)为富集的生物过程术语。此外,注释还揭示了分子功能(4861)、离子结合(2016)和氧化还原活性(767)等分子功能术语富集,以及细胞(2453)、细胞内(2352)和细胞组分(1972)等细胞组分术语富集(图1c)。 图1:C. pseudotenuipes基因组功能注释 病原体与宿主相互作用数据库(PHI库)注释结果表明,C. pseudotenuipes包含毒力降低(1108)、致病力未受影响(970)、致病性丧失(208)、致死(123)、高毒力(89)、植物无毒决定因子(16)、化学敏感性(7)、抗药性(5)和增强对抗性(2)的基因(图2)。结果表明,主要的注释基因是降低毒力和不影响致病性的,表明C. pseudotenuipes是一种轻度致病菌株。 图2:C. pseudotenuipes病原PHI表型突变类型的分布 此外,碳水化合物活性酶注释结果还显示C. pseudotenuipes含有396个相关基因,包括168个糖苷水解酶(GHs)和63个碳水化合物酯酶(CE)、56个辅助活性(AAs)、16个碳水化合物结合模块(CBM)和3个多糖裂解酶(PLs)(图3),此注释结果表明该菌株可能会捕获更多的能量并分解复杂的碳水化合物。 图3:C. pseudotenuipes CAZy酶功能分类 2. 比较基因组分析 将C. pseudotenuipes与三个近缘物种进行了比较基因组分析。结果表明,4个物种中基因组大小由大到小分别为:C. cicadae(34.11 Mb)、C. militaris(32.27 Mb)、C. pseudotenuipes(30.1 Mb)和C. tenuipes(30.06 Mb);scaffolds数依次为:C. cicadae(595个),C. pseudotenuipes(527个)、C. tenuipes(285个)和C. militaris(32个)。C. pseudotenuipes、C. tenuipes、C. cicadae和C. militaris的GC含量分别为54.11%、53.72%、52.70%和51.40%。 表2:虫草属基因组组装结果比较 3. 次生代谢产物合成基因簇分析 由次级代谢产物基因簇结果发现,C. pseudotenuipes和C. cicadae具有相同的SM BGC(31),其次是C. militaris(29)和C. tenuipes(28)。预测的BGC与MIBiG数据库中的已知簇显示出不同程度的遗传同源性,其中C. pseudotenuipes的同源性最高(37.93%),其次是C. tenuipes(28.57%)和C. cicadae(25.81%)。C. pseudotenuipes、C. tenuipes、C. cicadae和C. militaris基因组预测的BGC可能合成dimethylcoprogen。相比之下,通过C. pseudotenuipes、C. tenuipes和C. cicadae中预测的BGC预测了催化leucinostatin A/B、neosartorin、dimethylcoprogen wortmanamide A/B和白僵菌素等生物合成的基因。这些结果表明,同属的相似物种具有不同类型和数量的BGC及催化合成化合物。 C. pseudotenuipes、C. tenuipes和C. cicadae的几条BGC序列与MIBiG序列100%相似。预测的C. militaris区域358.1、C. pseudotenuipes区域71.1、C. cicadae区域56.2和C. tenuipes区域18.2可能与dimethylcodogen生物合成有关(图4a)。C. pseudotenuipes区域111.1和区域34.1负责epichloenin A的生物合成(图4b),C. pseudotenuipes区域2.2负责clavaric acid的生物合成。Local-BLAST比对结果表明,dimethylcodogen合成需要具有A-P-C-P-C结构域、乙酰转移酶、PRK08315超家族和MFS超家族的酶。此外,epichloenin A的合成需要NRPS(A-A-A-P-C)、PTZ00265超家族和MFS超家族。 图4:C. pseudotenuipes、C. tenuipes、C. militaris和C. cicadae的BGC与已鉴定的dimethylcoprogen(a)和epichloenin A(b)的生物合成BGC的成分比较 C. pseudotenuipes、C. tenuipes、C. cicadae和C. militaris中假定SM BGC的五个主要生物合成基因与MIBiG数据库中已知的基因簇相似(40%至85%)。C. pseudotenuipes 区域78.1、C. cicadae区域94.1和C. militaris区域426.1是鲨烯合成抑制剂S1的合成区。C. tenuipes区域38.2、C. cicadae区域117.1和C. militaris区域95.1可能合成viriditoxin,而C. cicadae区域1.2、C. militaris区域346.1和区域468.1分别合成铁色素、phomasetin和fumosorinone。结果表明, C. pseudotenuipes缺乏合成viriditoxin的潜力,催化合成viriditoxin除了需要核心酶PKS(SAT-KS-AT-PT-ACP-ACP-ACP-TE)外,还需要其他几种酶(图5a)。此外,C. militaris 区域346.1的核心基因结构域(KS-AT-MT-KR-ACP-C-A-P-TE)和修饰基因与LC361337.1相似。由于修饰基因的位置和方向不同,其余三个物种缺乏合成phomasetin的潜力。这些结果表明,C. militaris 区域468.1与合成fumosorinone的基因簇相似(图5c)。结果表明功能酶的位置和方向在物种之间不同。 图5:比较viriditoxin(a)、phomasetin(b)和fumosorinone(c)假定生物合成基因簇 C. tenuipes、C. cicadae和C. pseudotenuipes的整个基因组包含的基因簇与那些负责白僵菌素(BEA)BGC合成的基因簇高度相似(图6)。虫草属和球孢白僵菌之间的BEA BGC高度相似性表明,这些属之间存在一定程度的水平基因转移,尽管基因序列在方向和位置上有所不同,但在不同物种之间可能发生基因丢失/获得。 图6:比较白僵菌素生物合成基因簇和结构 此外,C. tenuipes区域36.1、C. cicadae区域27.1和C. pseudotenuipes区域112.1的基因簇类似于wortmanamide A/B BGC(图7)。C. tenuipes区域36.1、C. cicadae区域27.1和C. pseudotenuipes区域112.1的杂交PKS-NRPS结构域为KS-AT-DH-MT-KR-ACP-C-A-(P)-SDR。KS-AT-DH-MT-KR-ACP-C是催化合成wortmanamide A/B的结构域。这些结构域也有略微不同的修饰基因。因此,C. tenuipes区域36.1、C. cicadae区域27.1、C. pseudotenuipes区域112.1可能会合成wortmanamide或其类似物。 图7:比较wortmanamide A或wortmanamide B生物合成基因簇和结构 4. 聚类分析 虫草属PKS和PKS-NRPS与其他真菌PKS和PKS-NRPS的聚类结果表明,C. militaris区域346.1与Pyrenochaetopsis物种聚集,可能催化phomasetin或其类似物的生物合成。C. militaris区域95.1、C. cicadae区域117.1和C. tenuipes区域38.2与PKS蛋白聚集在一起,可能催化Paecilomyces variotii中的viriditoxin或其类似物合成。C. pseudotenuipes区域112.1,C. cicadae区域27.1、C. tenuipes区域36.1和邬氏黄丝曲霉分别聚集在一个独立分支上,可能催化wortmanamide A/B或其类似物的合成。C. militaris区域468.1与C. fumosorosea聚集在一起,可能催化fumosorinone或其类似物的生物合成。C. pseudotenuipes区域71.1、C. cicadae区域56.2和C. tenuipes区域18.2与交链格孢菌聚集在一起,可能产生dimethylcoprogen或其类似物。C.pseudotenuipes区域44.1、C. cicadae区域87.1和C. tenuipes区域63.1聚集在白僵菌的一个单独分支上,可能催化BEA或其类似物的生物合成。同样,C. pseudotenuipes区域111.1和C. tenuipes区域34.1与Epichloe festucae聚集在一个独立的分支上,是一段Epichlo festuceae中催化Epichlonein A生物合成的蛋白质序列。故假设这两个区域可能催化Epichlonein A或其类似物的合成。 5. 共线性分析 对含有SM BGC的C. cicadae(24)、C. militaris(26)、C. pseudotenuipes (27)和C. tenuipes(28)的基因组scaffold进行了共线性分析。SM BGC所在的scaffold分为70多个共线区块,存在重排现象(图8)。 图8:含有次生代谢物合成基因簇scaffold的共线性分析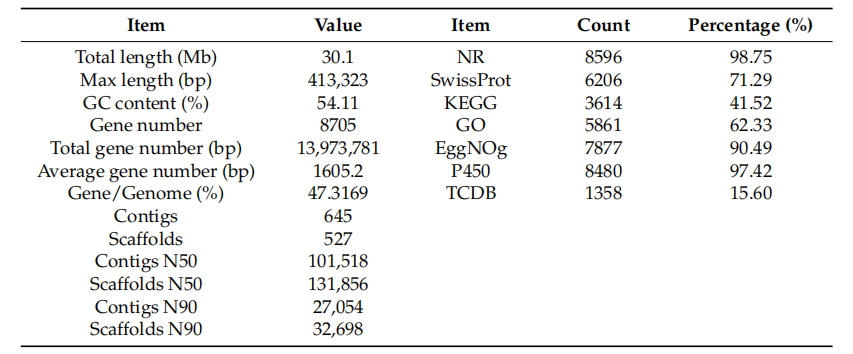



结论 本研究对C. pseudotenuipes基因组进行了测序分析,发现其基因组在调节蛋白活性和转化、信号转导和代谢、碳水化合物运输和代谢、捕获能量和分解碳水化合物等方面具有丰富的功能基因。此外,虫草属物种只有七个假定的BGCs与已知的基因簇高度相似,表明该物种具有产生其他次生代谢物的巨大潜力,这些发现为靶向基因组挖掘提供了可能。
本研究的denovo测序和部分分析由上海派森诺生物科技股份有限公司完成。如需进一步讨论,欢迎发邮件或者致电我们哟(邮箱地址:microsupport@personalbio.cn,联系电话:025-56165883-832)!
文章索引:Lu Y, Wang Y, Yuan X, et al. Genomic Comparative Analysis of Cordyceps pseudotenuipes with Other Species from Cordyceps. Metabolites 2022, 12, 844.