
2022-11-07

《Molecular Ecology》 影响因子:6.622
近日,新西兰怀卡托大学科学院在生物领域的《Molecular Ecology》发表新研究成果!本研究通过对各国茶翅蝽品种进行ddRAD-Seq,获取差异SNP位点,结果表明变异位点相关基因具有杀虫剂抗性和感官感知功能,这为未来的研究与管理奠定了基础。
研究背景
茶翅蝽是一种高度侵袭性的害虫,原产于东亚,是一种多食性昆虫。它具有很高的扩散能力和与特有物种竞争的潜力,给农业和园艺造成严重的滋扰。1996年,茶翅蝽首次在宾夕法尼亚州阿伦顿的本土范围外被报道,此后在美国、加拿大、智利和欧洲的其他地区出现。单基因座或全基因组SNP数据揭示了东亚土著群体之间高度不同的谱系。中国被认为是最有可能的全球传播源,有证据表明中国境外多次入侵,同时也发现了从美国到欧洲和智利的桥头堡事件。尽管广泛使用杀虫剂、信息素诱饵和植物气味诱饵,茶翅蝽仍然对农业造成损害。本研究利用ddRAD-seq发现了与杀虫剂抗性和嗅觉基因非常接近的显著差异SNP,比较群体中异常SNP等位基因频率的差异表明,茶翅蝽入侵成功可能是由长期遗传变异演变而来的。尽管迄今为止没有茶翅蝽杀虫剂抗药性的记录,但研究结果表明,这些性状的潜在进化能力很强,这突出了未来可持续和有针对性的管理战略的必要性。
研究材料与方法
1、实验材料
各国茶翅蝽(新西兰,中国,意大利,美国等) 2、测序平台 Illumina HiSeq X Ten 3、分析内容 PCA分析,群体结构分析,基因流分析等。
研究结果
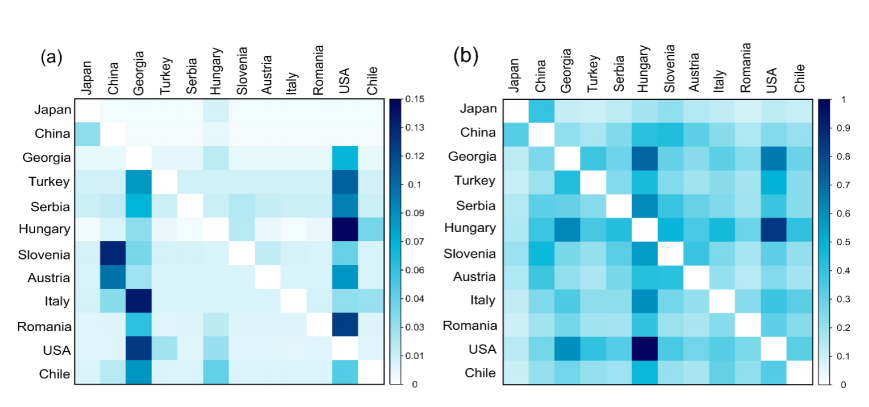
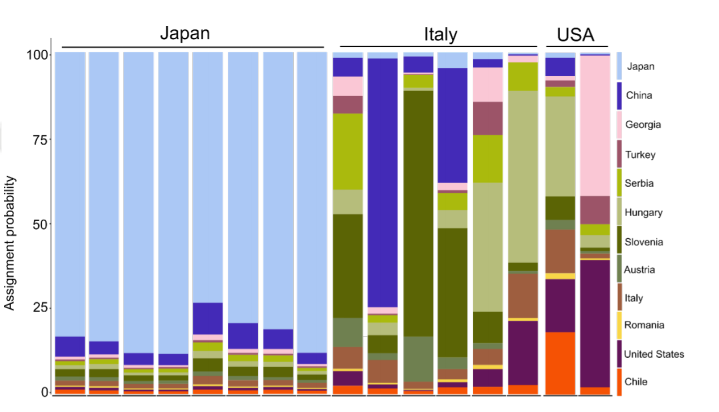
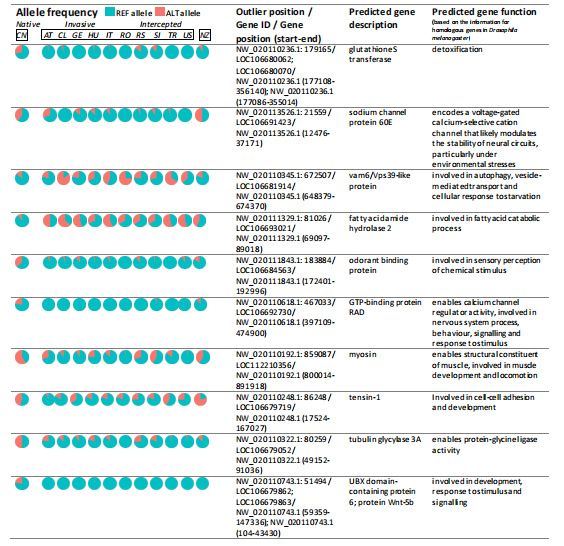
1. 群体分化 基于除新西兰截获的样本外,从2个原住国和10个入侵国家获得的404个样本的14,172个SNP分析显示,在日本和所有入侵国家之间观察到最高的FST(为0.0519-0.107)。然而中国与入侵种群之间的FST相对较低(为0.0189-0.0646)。在入侵种群中,罗马尼亚样本高度分化,FST值最大(图1b)。空间基因组分析显示,日本样本是一个高度分化的群体,来自中国的样本更接近入侵种群(图1c)。PC1分布表明,中国样本与侵入性PC空间右侧边缘的侵入性样本重叠,但侵入性群体的跨度更大,这表明侵入性群体的基因组变异水平更高。PC2表明,日本和中国之间的遗传距离小于日本和入侵群体之间的距离。六个来自中国的样本显示出与日本有共同血统的特征-它们在PCA中与这两个国家的距离大致相等(图1c)。 群体结构分析结果与PCA一致:中国和日本形成了单独的集群,以及在不同k值下中国和所有入侵种群之间显著混合的一致信号(图1d)。来自中国的六个离群样本与日本有着特殊的混合(血统比例>0.5)。在已确定的入侵种群中,奥地利和斯洛文尼亚与中国的混合率最高,而罗马尼亚与其他入侵种群相比差异很大(图1d)。表明中国和日本是两个不同的谱系,并表明中国种群和入侵种群之间有密切的系统发育关系。 图1 茶翅蝽的全球基因组结构 2.基因交流 群体迁移率结果表明,当代有效基因流动率从本土范围到入侵范围都很低,反之亦然(平均后验分布<0.01),但在大多数入侵国家中都很高(从0.01到0.15,特别是从不同的入侵国家到美国和格鲁吉亚(图2a)。DivMigrate结果显示了所有种群之间的相对迁移,而中国和入侵种群之间的迁移率较低(GST<0.5)(图2b)。日本与所有入侵种群之间的迁移率也很低(GST介于0.089-0.216之间)。在入侵地区中,从意大利、美国、塞尔维亚、斯洛文尼亚和格鲁吉亚观察到高相对迁移率(GST>0.5),以及从匈牙利、土耳其和格鲁吉亚移民到美国(图2b)。 图2 茶翅蝽不同种群间当代基因流动模式与定向相对迁移率的比较 3.截获种群的来源预测 评估了在新西兰边境截获的来自意大利、美国和日本的集装箱中的16个群体样本,以确定我们是否能够正确识别每个样本的来源。assignPOP方法在确定原产样本的来源种群方面显示出更高的准确性,从日本截获的所有八个茶翅蝽样本均以高后验概率正确分配(后验概率>0.7;图3)。然而,匈牙利和格鲁吉亚被错误地认定为从美国运出的货物中发现的新西兰截获样本的最可能来源(尽管后验概率很低:0.29和0.41)。同样,在意大利货物中发现的六个样本未被分配给斯洛文尼亚、中国和匈牙利的来源种群,每种情况下的分配后验概率为中高(后验概率范围为0.3-0.72;图3)。 图3新西兰种群的来源预测 4.受到选择的SNP位点和基因 通过对中国入侵种群与本土种群进行全基因组选择清除分析获得63个异常SNP,除了两个SNP共同位于同一scaffold上的个例外,其余异常点位于不同的scaffold上。变异注释显示,约24%、15%和1.5%的SNP分别位于基因组的转录区、上游或下游基因区和外显子区,其余的位于基因间区(约36%)或内含子(约23%)。使用UCSC Table Browser提供的茶翅蝽基因组注释了这些外显子的55个独特转录物。一些相关基因的产物在入侵中具有重要性的潜在适应性,包括杀虫剂抗性、解毒、脂肪酸代谢和感官感知(表1)。GO富集分析显示,这55个独特转录物显著富集在包括氧化还原酶活性(GO:0016491)、气味结合(GO:0005549)和蛋白质结合(GO:0005515)以及一种生物过程(细胞蛋白质修饰过程;GO:0006464)的通路(p<0.05)。 表1 从本地和入侵群体的基因组中扫描得到的10个关键基因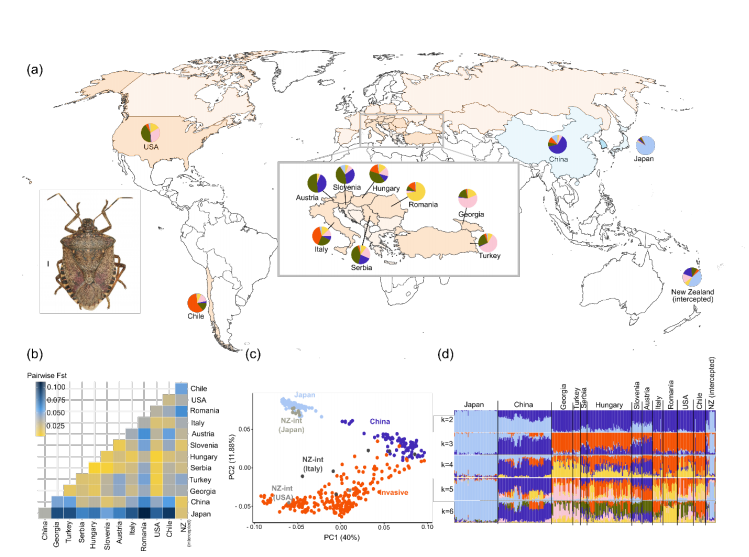
结 论
全基因组SNP数据使我们能够描述高度多食性害虫的全球入侵模式。茶翅蝽的复杂入侵历史包括多个殖民事件、高水平的基因交流以及引入种群之间的混合,导致无法将多个入侵准确地分配给源种群。全基因组数据可以为这些最近的扩散和混合事件提供更精细的区分,从而允许在入侵物种引入范围内没有离散种群的情况下进行种群分配。此外,我们确定了可能与成功入侵相关的具有生物学意义的基因组变异,变异位于基因附近,这些基因具有众所周知的杀虫剂抗性和感官感知功能。尽管目前还没有关于茶翅蝽杀虫剂抗性的报道,但本结果表明,抗性表型可能为长期变异中进化累计获得。
本研究的ddRAD-seq由上海派森诺生物科技股份有限公司完成。如需进一步讨论,欢迎发邮件或者致电我们哟(邮箱地址:genome_support@personalbio.cn,联系电话:025-56165883-834)!
文章索引: Elahe Parvizi, Manpreet K. Dhami, Juncong Yan, et al. Population genomic insights into invasion success in the polyphagous agricultural pest, Halyomorpha halys [J]. Molecular Ecology, 2022. doi: 10.1111/mec.16740.