
2022-12-26

《Journal of Fungi》
影响因子:5.724
近日,吉林大学在微生物领域的《Journal of Fungi》发表新研究成果!本研究将葡萄中分离得到的非酿酒酵母菌S.bacillus CC-PT4 (CGMCC No. 23573)进行基因组测序和生物信息学分析,分析其有益特性、次生代谢产物、生物合成基因簇、杀伤毒素、溶葡萄球菌素、胁迫适应性、絮凝性和粘附安全性,为后续该菌株的研究奠定基础。
01、研究背景
非酿酒酵母常见于葡萄、酿酒及相关环境中。该物种于2002年首次在美国纳帕谷受葡萄霉变影响的葡萄酒中分离,随后在匈牙利的白葡萄酒中分离,鉴定其为新种并命名为Candida zemplinina。经同工酶谱、26S rDNA酶切谱、26S rDNA测序分析,最终归类为S. bacillaris 。S.bacillus具有良好的高渗透耐受性,可以耐受高浓度的糖和乙醇,该菌株在发酵过程中产生甘油和低浓度乙醇以及多种风味。此外,S.bacillus具有嗜果糖特性,Saccharomyces cerevisiae具有嗜糖特性,这使得它们能够在发酵过程中长时间共存。许多研究发现,酵母与酿酒酵母混合发酵有利于提高葡萄酒的香气。
化学杀菌剂是抑制病原体的传统方法,但反复使用这些化合物通常会导致各种不良反应。然而,生物防治剂是减少化学杀菌剂使用的一种潜在替代品。酿酒酵母、布拉尔氏酵母和马氏克鲁维酵母都已被证明具有生物防治作用,杆菌也被证明具有生物防治的潜力。其抑菌机制可能包括营养物竞争、空间竞争、产生降解酶、杀伤毒素和挥发性有机物等。本文对S.bacillus CC-PT4进行了全基因组测序并进行了生物信息学分析,发现S.bacillus CC-PT4对MRSA(抗药性金黄色葡萄球菌)具有抑制作用,对恶劣环境具有良好的适应性。此外,S.bacillus CC-PT4携带与致病性和耐药性相关的基因。总的来说,本研究对S.bacillus CC-PT4的全基因组测序和分析为了解该菌株的生物学特性和进一步开发提供了有价值的信息。
02、 研究材料与方法
1.实验材料:S.bacillus CC-PT4
2.测序平台:Illumina NovaSeq + PacBio
3.分析内容:真菌近完成图,次生代谢产物合成基因簇预测,絮凝与粘附分析等。
03、研究结果
1. S.bacillus CC-PT4基因组测序及组装
S.bacillus CC-PT4全基因组大小为9.45 Mb, GC含量为39.50%。包含5个scaffolds,最长scaffold为4,159,713 bp,最短scaffold为8,503 bp, N50为4,154,241 bp。S.bacillus CC-PT4基因组圈图如图1所示。通过在基因组序列中完全对齐的单拷贝基因数量占单拷贝基因总数的百分比来评估基因组组装的完整性,其中63.3%的基因与BUSCO单拷贝基因完全对齐。
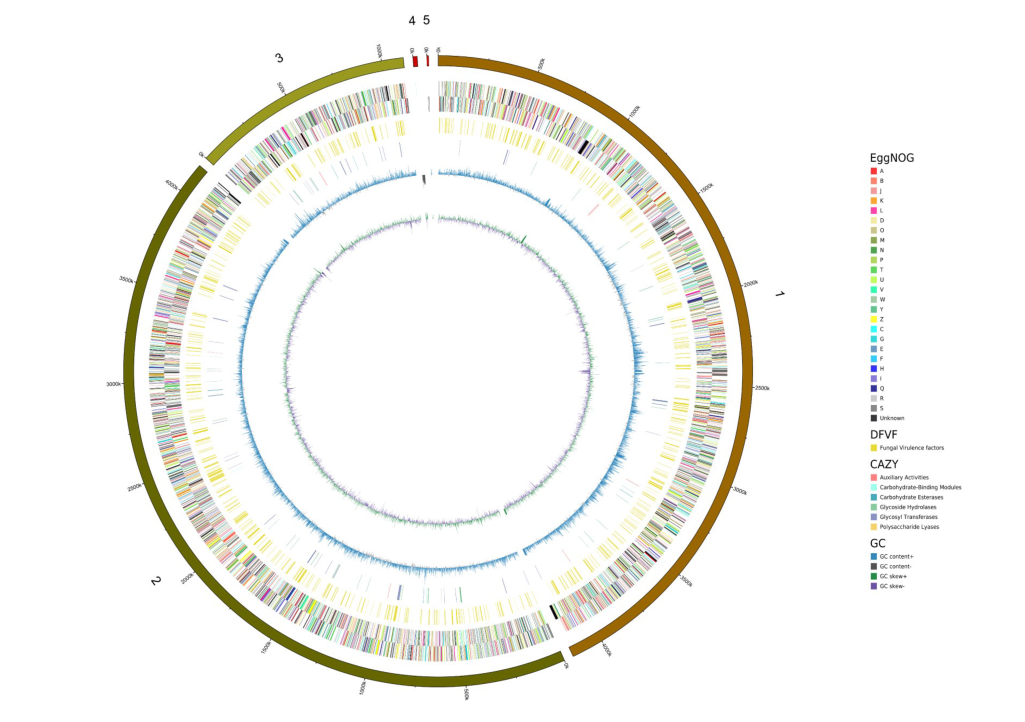
图1 S.bacillus CC-PT4基因组圈图
2.功能原件分析
经预测,S.bacillus CC-PT4有286个串联重复序列和1,020个分散在基因组中的穿插重复序列,S.bacillus CC-PT4有123个tRNAs, 4个rRNAs和7个其他非编码RNAs。菌株CC-PT4基因组共预测到4,150个基因,占总基因组长度的63.70%,平均每个基因长度为1450.8 bp,共有4,266个外显子,平均每个基因有1个外显子,占总基因组长度的63.44%。
3. 基因注释
在蛋白编码基因中,P450数据库共注释了4,056个(97.73%)基因,其次是NCBI nr数据库(3,538个,5.25%)、eggNOG数据库(3,185个,76.45%)、KEGG数据库(2,610个,62.89%)、Swiss-Prot数据库(3,363个,81.04%)、Go数据库(2,888个,69.59%),TCDB数据库(670个,16.14%)。
本研究统计了S.bacillus CC-PT4在NCBI nr中注释到的3,538个基因的物种信息,排名前25位的物种共涉及3,074个基因(图2),但标注的基因中有10个序列一致性不低于97%的基因属于Starmerella属,9个基因为S.bacillus,其中4个基因序列一致性为100%(表1)。
进一步,用eggNOG对S.bacillus CC-PT4蛋白进行功能注释并进行聚类分析。用eggNOG对S.bacillus CC-PT4标记的3,185个基因的分类结果如图所示。有499个基因没有明确的功能,这可能与缺乏对杆菌的研究和缺乏内参基因有关。功能分类明确的注释基因最多的是翻译、核糖体结构和生物发生(321个);与碳水化合物转运代谢、脂质转运代谢、氨基酸转运代谢相关的基因分别有146、108、145个。而细胞外结构和核结构所标注的信息较少(4个基因)。有57个基因与次生代谢产物的生物合成、转运和分解代谢相关。此外,未发现与细胞运动相关的基因,这与酵母是一种非运动微生物的事实是一致的。
KEGG共标记2,610个基因,分为8个大类和50个亚类。其中,与遗传信息处理功能相关的基因数量最多,其次是信号转导和细胞过程。Swiss-Prot数据库中S.bacillus CC-PT4基因组注释到3,363个基因。在GO数据库中,S.bacillus CC-PT4基因组有4,109个分子功能注释基因,8,560个细胞成分注释基因,10,108个生物过程注释基因。
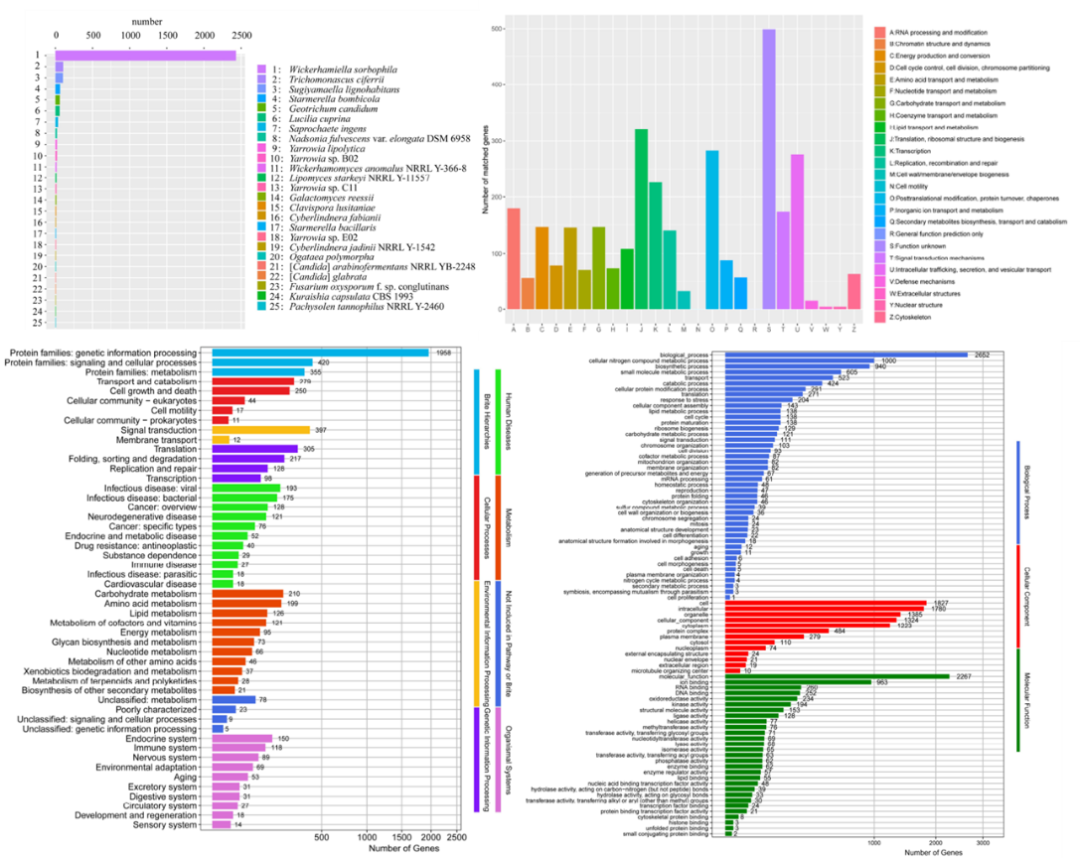
图2 各数据库注释统计结果

表1 NCBI nr注释的S.bacillus CC-PT4基因组序列统计(前10)
4. CAZy分析
CAZy数据库主要包含与糖苷键的降解、修饰和生成相关的酶家族。酶家族被分为六类:糖苷水解酶(GHs)、多糖裂解酶(PLs)、碳水化合物酯酶(CEs)、糖基转移酶(GTs)、辅助活性(AAs)和碳水化合物结合模块(CBM)。经预测,S.bacillus CC-PT4含有95个CAZy酶基因(表2)。
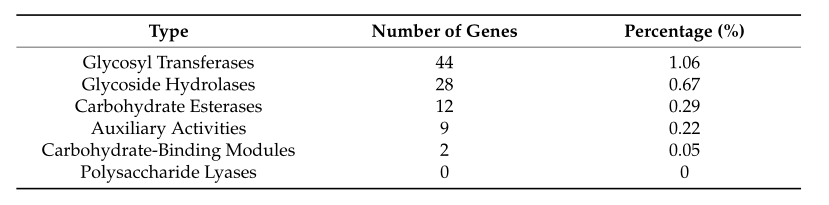
表2 CAZy分析统计
5.次生代谢产物合成基因簇
在antiSMASH预测的S.bacillus CC-PT4基因组中发现了两个次生代谢产物生物合成基因簇,分别是非核糖体肽合成酶簇和萜烯类。对上述基因进行MIBIG数据库注释,各基因BLAST得分最高的注释结果见表3。
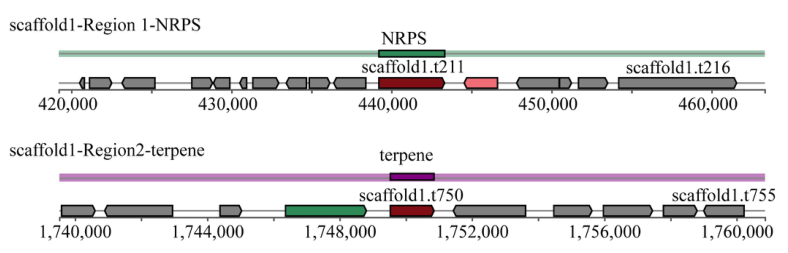
图3 S.bacillus CC-PT4次生代谢产物生物合成基因簇的预测结果
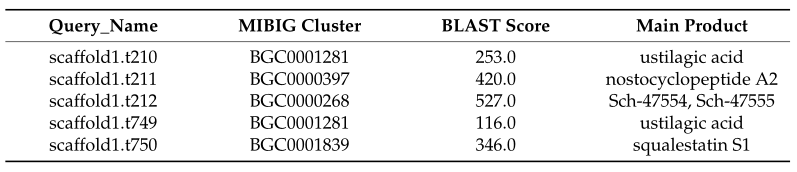
表3 基于MIBIG的S.bacillus CC-PT4次级代谢合成基因簇的注释结果
6. Killer toxin 和 Lysostaphin基因
Killer toxin是酵母分泌的一种有毒蛋白质,可以杀死其他酵母,但对自己不会造成伤害。Lysostaphin可特异性水解金黄色葡萄球菌肽聚糖的五甘氨酸交联,从而裂解金黄色葡萄球菌,对耐药金黄色葡萄球菌具有同样的抑菌作用。根据S.bacillus CC-PT4基因组的注释结果显示有2个基因编码Killer toxin,1个基因编码Lysostaphin (表4)。

表4 S.bacillus CC-PT4基因组中的Killer toxin 和 Lysostaphin基因
7. 适应力和耐药分析
根据酵母基因组在eggNOG、Swiss-Prot和NCBI nr数据库中的注释,寻找参与胁迫适应的相关基因。结果表明,S.bacillus CC-PT4基因组中存在许多有利于菌株适应恶劣胁迫的基因,包括抗pH胁迫、抗胆汁胁迫、抗氧化应激、抗离子和重金属胁迫、抗热应激和其他胁迫。根据S.bacillus CC-PT4基因组的注释结果,该菌株中有30个耐药相关基因。
8. ,絮凝与粘附性分析
从SGD下载FLO1、FLO5、FLO8、FLO9、FLO10、FLO11、FIG2和AGA1编码絮凝蛋白序列,并对S.bacillus CC-PT4基因组进行BLAST处理。结果表明,S.bacillus CC-PT4基因组中有5个基因(scaffold1.378, scaffold1.1051, scaffold1.1055, scaffold2.1852, scaffold2.1859)编码絮凝蛋白。从图4可以看出,基因scaffold1.378与FLO1、FLO5、FLO10三个絮凝蛋白具有序列一致性,基因scaffold2.1852与FLO1、FLO5、与FLO9中的具有序列一致性,基因scaffold2.1859与FLO9、FLO10具有序列一致性。此外,这些基因与絮凝蛋白的多个序列片段具有同源性。
此外,从SGD中下载了41个与粘附相关的蛋白序列,并利用BLAST对S.bacillus CC-PT4基因组和这些蛋白进行了分析。结果显示,S.bacillus CC-PT4基因组中65个基因与粘附蛋白BGL2、CKA2、CRH1、CRR1、CWH41、DCW1、DFG5、FKS3、GSC2、KRE6、KTR1、LAS21、PST1、ROT2、SCW10、SCW11、SCW4、SKN1、SMK1、SPI1、SUN4、UTR2、YPS1、YPS3的一个或多个蛋白序列一致。
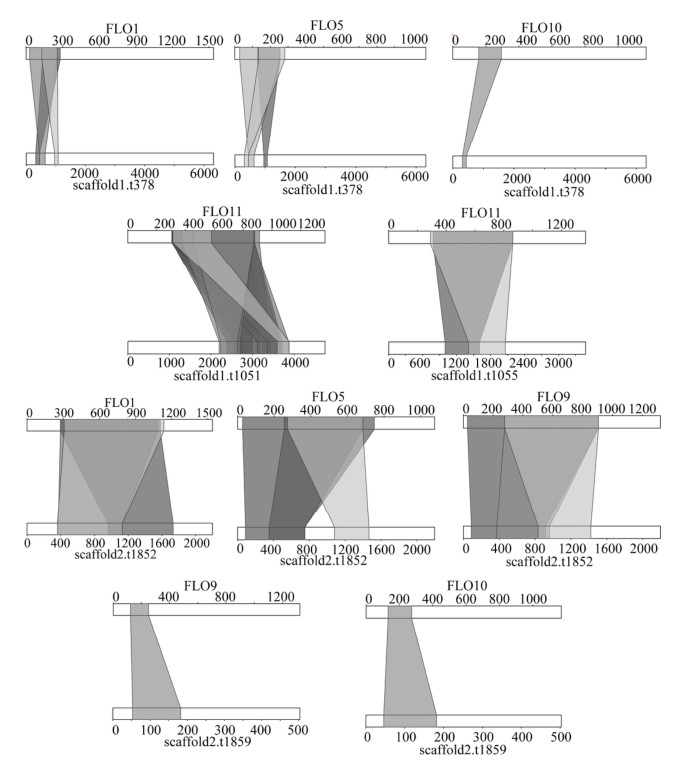
图4 利用絮凝蛋白序列对S.bacillus CC-PT4基因组进行BLAST分析
9. 致病性分析
根据S.bacillus CC-PT4全基因组预测的PHI表型突变型基因数量统计如图5所示。PHI数据库共注释到1013个基因,其中最多的是毒力降低,有545个基因,而拮抗增强的基因为零。通过进一步分析,发现与人类疾病相关的基因有34个(表5)。
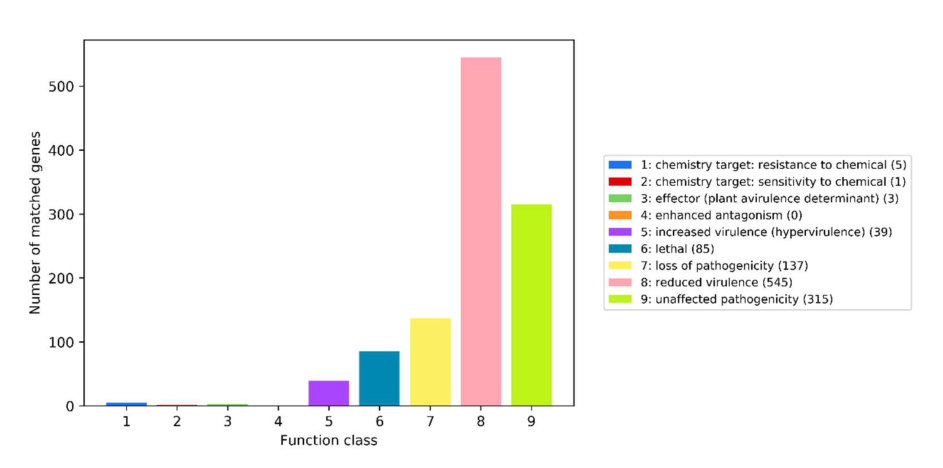
图5 S.bacillus CC-PT4中病原菌-宿主相互作用(PHI)基因的分布
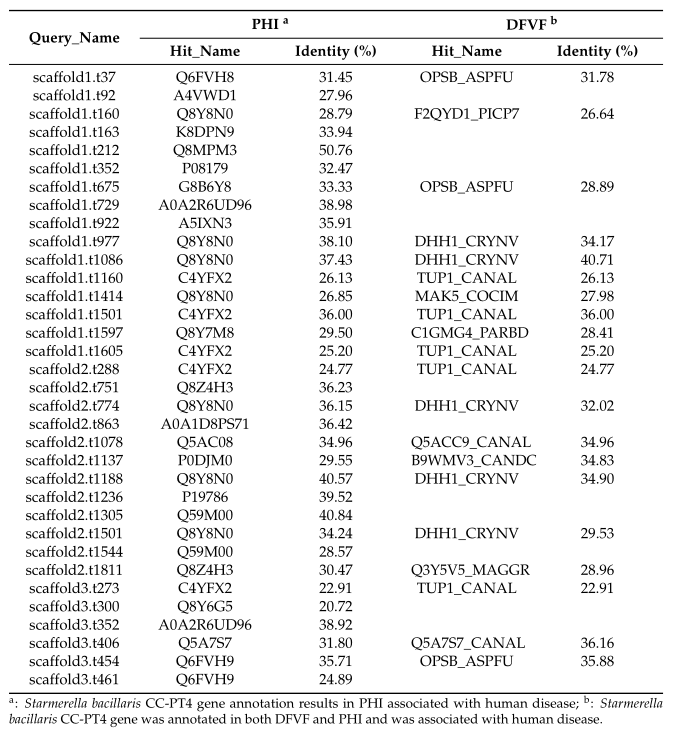
表5 S.bacillus CC-PT4毒力分析
04、结 论
本研究对S.bacillus CC-PT4的全基因组序列进行了组装和生物信息学分析。S.bacillus CC-PT4全基因组大小为9.45 Mb, GC含量为39.50%。此外,利用多个生物信息学数据库预测和注释了4150个蛋白质编码基因。蛋白质编码基因的注释结果显示,许多基因与应激适应、次生代谢产物、抗菌功能、安全性等相关,包括2个次生代谢产物合成基因簇,2个基因编码Killer toxin,1个基因编码Lysostaphin。综上所述,获得S.bacillus CC-PT4的全基因组序列有助于更好地了解该菌株的特性,有利于该菌株的挖掘和应用。
本研究的真菌基因组测序与分析由上海派森诺生物科技股份有限公司完成。如需进一步讨论,欢迎发邮件或者致电我们哟(邮箱地址:microsupport@personalbio.cn,联系电话:025-56165883-832)!
文章索引:
Yong Shen, Xue Bai, Xiran Zhou, et al. Whole-Genome Analysis of Starmerella bacillaris CC-PT4
against MRSA, a Non-Saccharomyces Yeast Isolated from Grape [J]. Journal of Fungi, 2022, 8:1255.
doi:10.3390/jof8121255