2023-02-13

《Nature Communications》
影响因子:17.694
碱基编辑器的胞嘧啶和腺嘌呤编辑分别需要胞嘧啶和腺苷脱氨酶,没有一种脱氨酶能够同时进行胞嘧啶和腺嘌呤编辑。此外,胞苷和腺苷脱氨酶的不同属性易导致各种类型的脱靶效应,包括对胞嘧啶碱基编辑器(CBE)的cas9独立DNA脱靶效应和对腺嘌呤碱基编辑器(ABE)特别严重的RNA脱靶效应。本研究表明,25个TadA同源基因可以通过单或双突变产生功能性ABEs、CBEs或ACBEs,这些同源基因显示出最小的cas9独立DNA脱靶效应和遗传毒性,其中B5ZCW4、Q57LE3、E8WVH3、Q13XZ4和B3PCY2是有希望进一步改造的候选同源基因。此外,通过额外的单碱基突变,可以进一步降低甚至消除TadA直系同源碱基编辑器的RNA脱靶效应。
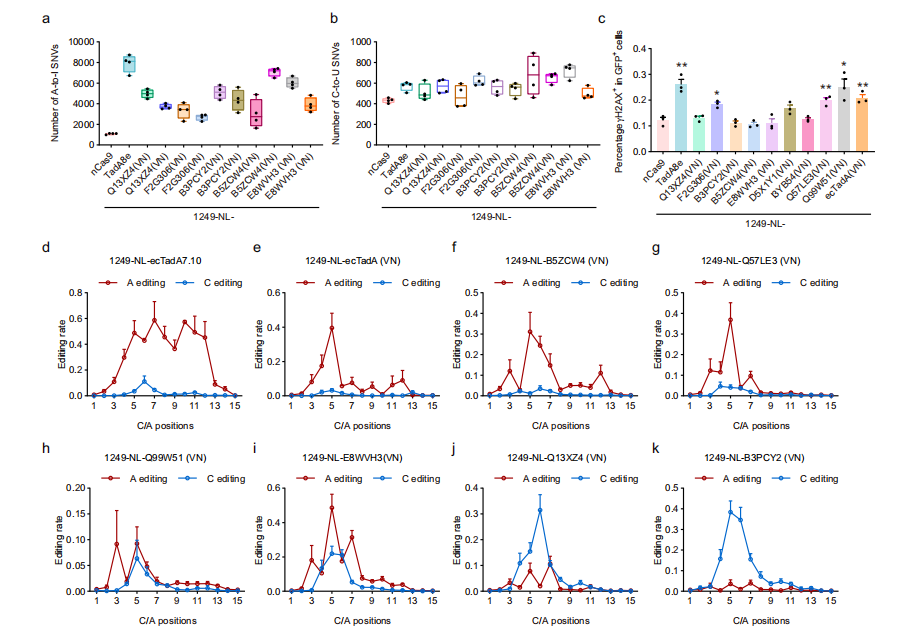
研究材料与方法 1.项目类型:扩增子测序 2.测序平台:Sanger、Illumina NovaSeq 3.分析内容:TadA同源基因的系统发育比较、TadA同源物的筛选与工程研究、R-loop检测cas9独立的DNA脱靶分析等。 图1 功能ABEs、CBE和ACBE的TadA同源性鉴定 图2 脱靶效应、具有代表性的TadA同源物衍生的碱基编辑器的遗传毒性和编辑范围 本研究对近千种TadA同源蛋白构建了系统进化树,并挑选>50种其它物种来源的TadA同源蛋白进行类似的改造和功能筛选。研究人员发现大肠杆菌来源的野生型ecTadA添加5’-NLS和3’-柔性接头序列并融合于nCas9内部的DS1249位点后,仅通过A106V和D108N双点突变便可获得具有明显腺嘌呤和胞嘧啶编辑活性的功能性碱基编辑器1249-NL-ecTadA(VN),其A5的A-G突变率为83%,C4的C-T突变率为27%。 总而言之,本研究通过筛选和设计TadA同源蛋白质工程化改造,证实了ABE、CBE以及ACBE在内的各类碱基编辑器开发的可行性,并减少或最小化了脱靶效应。本研究结果不仅扩展了碱基编辑的工具包,而且为编辑脱氨酶的潜在演化过程提供了重要的证据,同时也为碱基编辑器的开发拓展了新思路。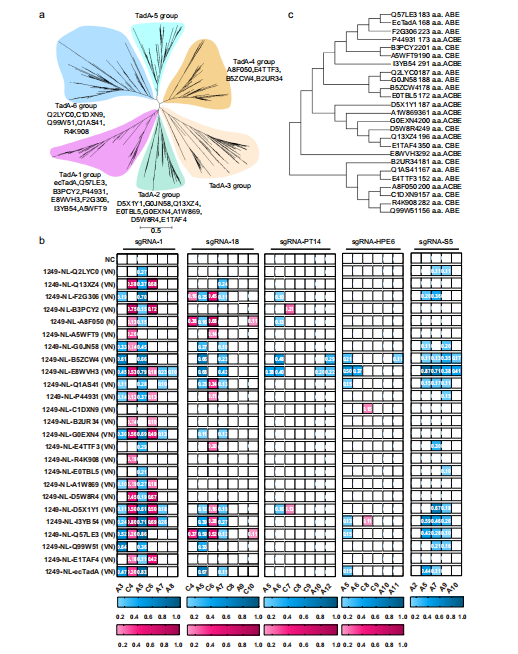
《Nature Communications》 影响因子:17.694
虽然微型CRISPR-Cas12f系统最近被开发出来,但衍生的微型胞嘧啶和腺嘌呤碱基编辑器(miniCBEs和miniABEs)的编辑功效和靶向范围尚未得到全面解决。此外,功能型miniCBEs尚未建立。本文,研究人员生成了各种Cas12f衍生的miniCBEs和miniABEs碱基编辑器,开发出功能性的miniCBEs和具有全新活性窗口、多样化的miniABEs。
研究材料与方法 1.项目类型:扩增子测序 2.测序平台:Illumina NovaSeq 3.分析内容:Cas独立的DNA脱靶效应分析、全基因组测序和数据分析、RNA编辑分析等。 图3活性高、范围多样的功能性d12抗体的开发 研究人员揭示了用传统胞苷脱氨酶生成的miniCBEs具有更大编辑窗口和高脱靶效应。为了改进经典miniCBEs和衍生miniABEs的编辑效率,研究人员对TadA脱氨酶进行诱变筛选,以高精度生成有效的miniCBEs,并将off-target效应降至最低。研究人员发现,新设计的miniCBEs和miniABEs能够纠正细胞系中的致病突变,并通过体内大脑中的腺相关病毒递送有效地引入基因突变。 总之,本研究有力地扩展了基础编辑工具包,并为广泛的遗传疾病提供了更多的治疗选择,提高了编辑活性。本研究还证明了TadA重编程为碱基编辑优化提供了另一种策略。本研究为CBE的发展提供了替代策略,扩展了miniCBEs和miniABEs的工具包,并为临床应用提供了有前途的治疗工具。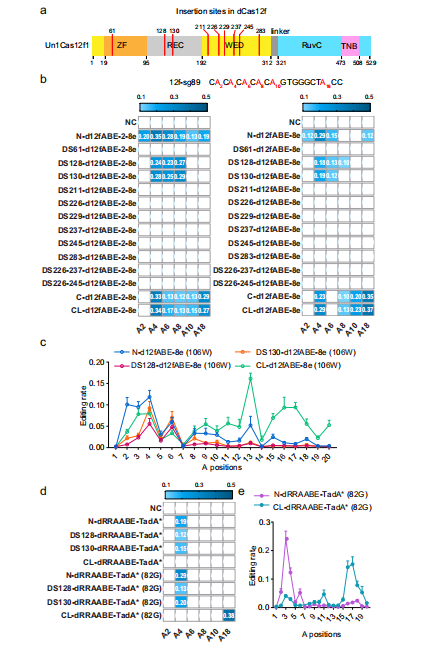
本研究的基因编辑序列测序由上海派森诺生物科技股份有限公司完成。如需进行基因编辑序列测序分析,欢迎发邮件或者致电我们哟(邮箱地址:genome_support@personalbio.cn,联系电话:021-80118168-6611)!
文章索引: [1]Zhang, S., Yuan, B., Cao, J. et al. TadA orthologs enable both cytosine and adenine editing of base editors. Nat Commun 14, 414 (2023). https://doi.org/10.1038/s41467-023-36003-3. [2]Zhang, S., Song, L., Yuan, B. et al. TadA reprogramming to generate potent miniature base editors with high precision. Nat Commun 14, 413 (2023). https://doi.org/10.1038/s41467-023-36004-2.


