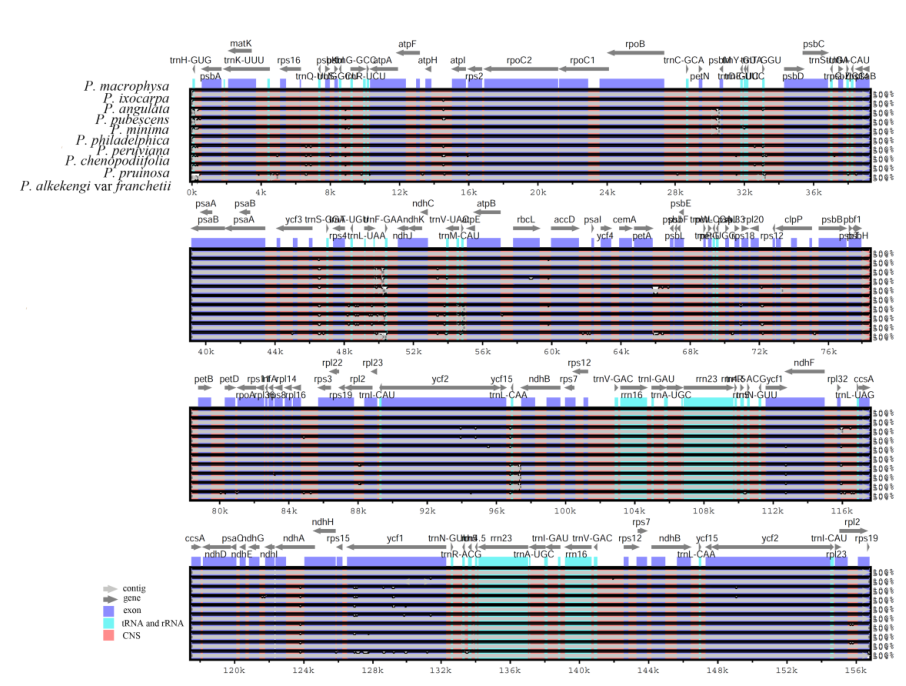
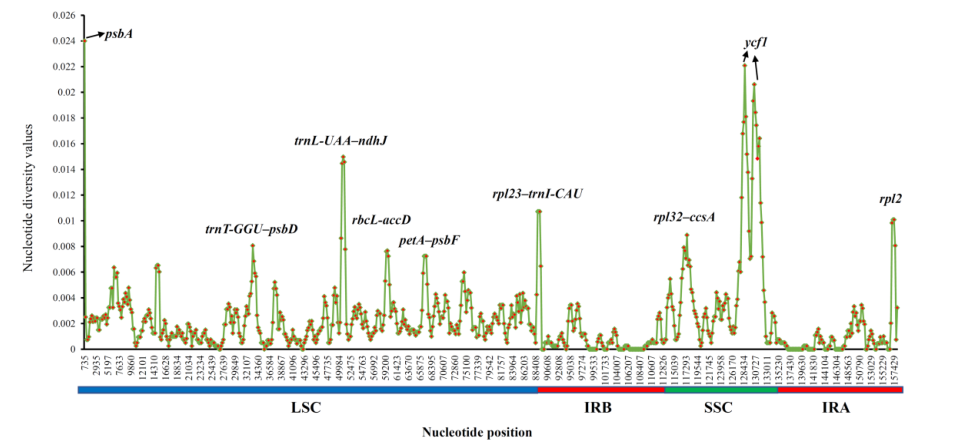
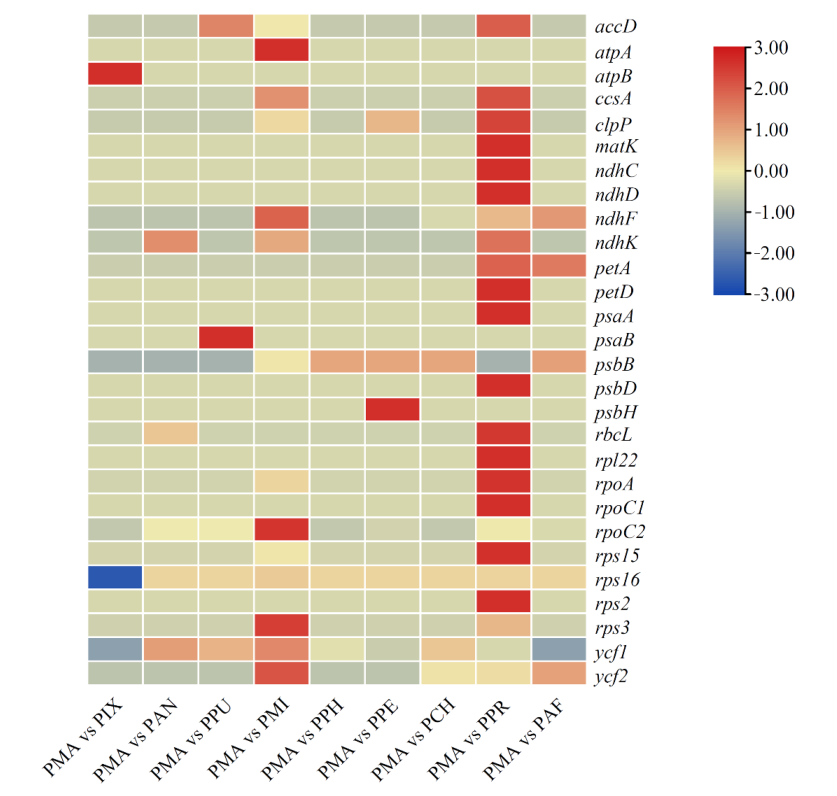
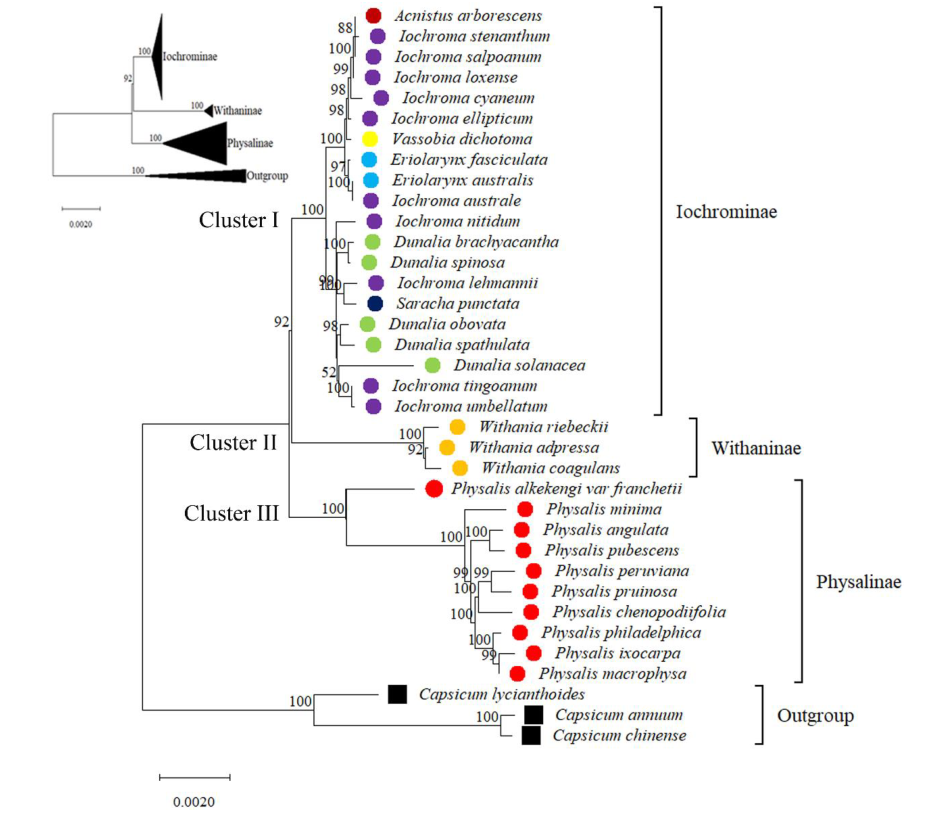
2023-05-22

《agronomy》
影响因子:3.949
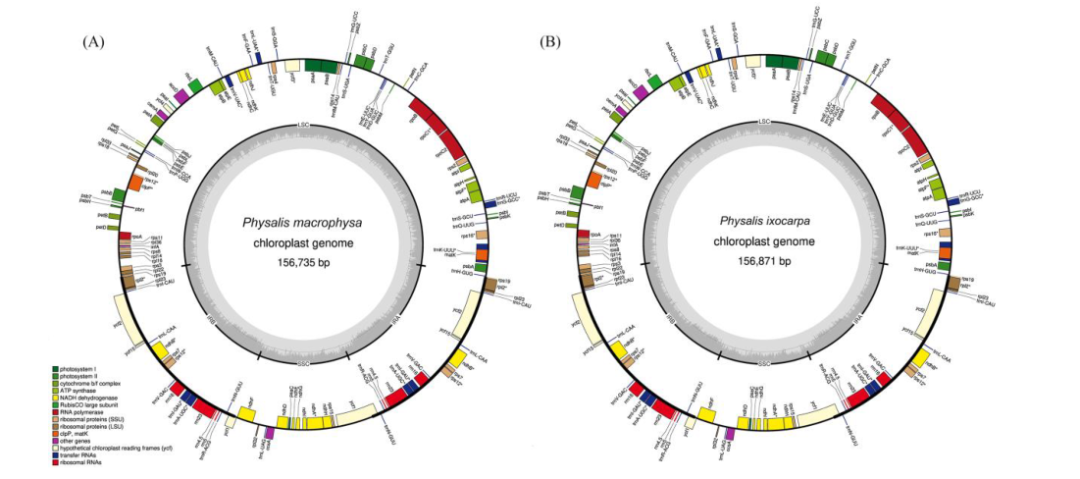
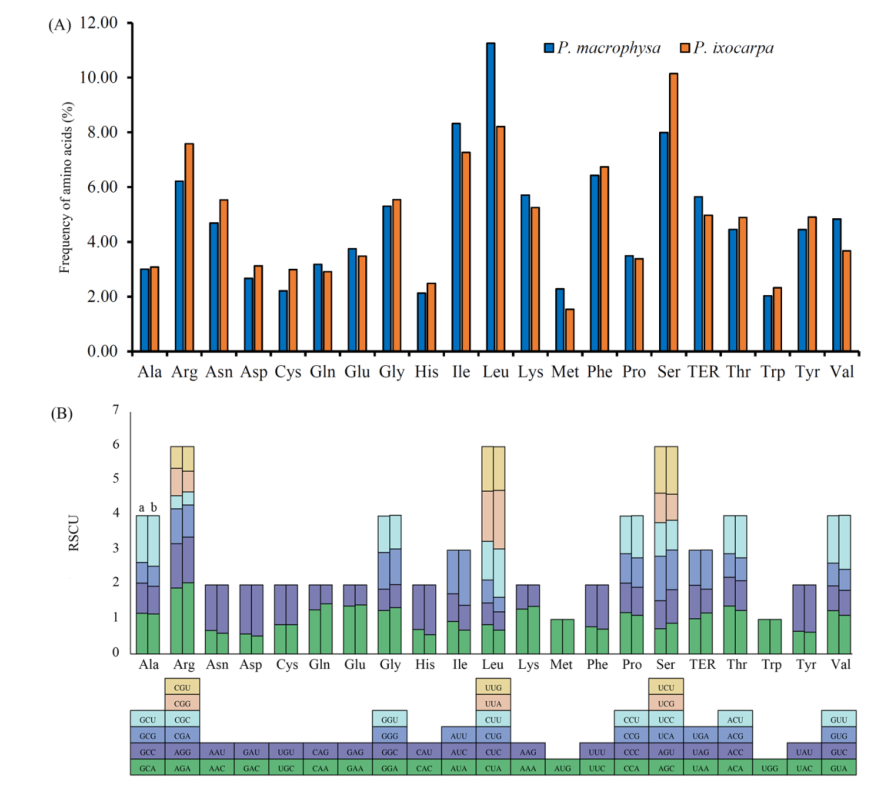
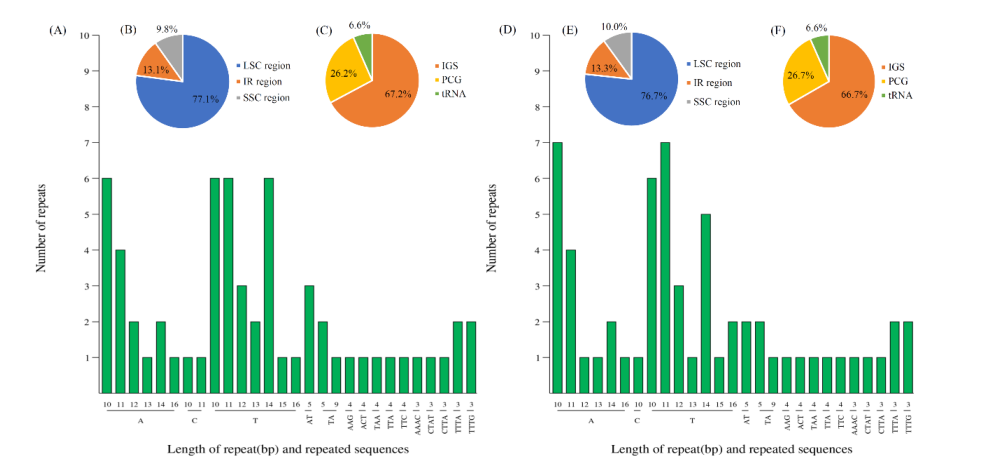
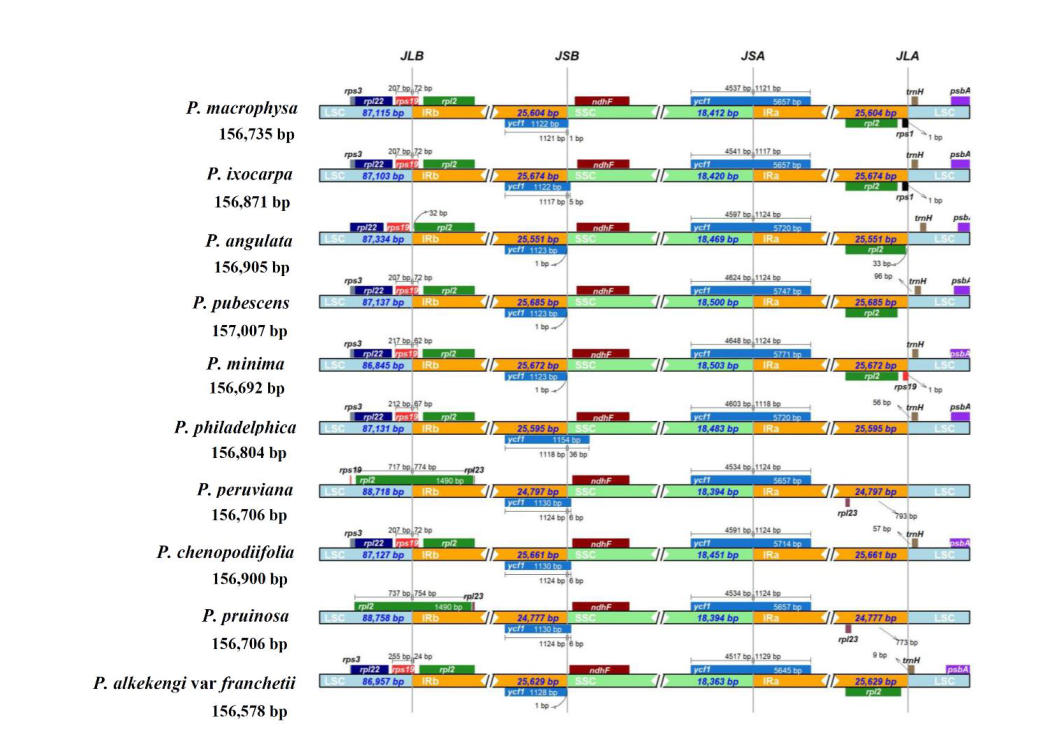
近日,杭州师范大学在农业和生物科学领域的《agronomy》发表新研究成果!本研究获得Physalis macrophysa (PMA)和Physalis ixocarpa (PIX)的叶绿体全基因组序列,并与8个酸浆属的叶绿体基因组进行了比较。本研究对两种酸浆属的叶绿体基因组进行分析,将有助于进一步研究酸浆属的物种鉴定、系统发育、进化以及种质资源开发潜力。 研究背景 酸浆属是茄属植物中最大的一个属,由大约90种组成,其中大多数具有重要的经济、药用或观赏价值的经济、药用或观赏价值。传统上,physaleaceae的物种是根据形态特征进行分类的。然而,该部落成员的形态特征相对相似,这为该部落几个属之间的系统发育关系鉴定带来了极大的困难。有研究结果表明,叶绿体基因组可以提供大量的遗传信息,可用于植物物种鉴定、DNA标记的开发以及叶绿体基因组水平上的分类和系统发育关系的解决。本研究选取了形态特征存在明显差异且生境不同的PMA和PIX进行研究,以扩大Physalis属质体基因组资源,并以此为基础研究Physalis属叶绿体基因组变异。 研究材料与方法 1.实验材料:Physalis macrophysa (PMA)和Physalis ixocarpa (PIX) 2.测序平台:Illumina 3.分析内容:叶绿体基因组denovo测序、密码子偏好性分析、重复序列分析、mVISTA分析、Ka/Ks分析、IR收缩与扩张分析、进化树构建等。 研究结果 叶绿体基因组特征 PMA的cp基因组大小为156,735 bp,PIX的cp基因组大小为156,871 bp。两个cp基因组呈现典型的四部结构,包括一个LSC区,一个SSC区和一对IR区。两个cp基因组包含相同的132个基因,编码87个蛋白质、8个rRNA和37个tRNA。对所有基因进行注释,并将其分为光合作用基因、自复制相关基因、功能未知基因和其他基因。 图1:PMA和PIX基因组圈图 密码子偏好性分析 蛋白质编码基因在PMA和PIX的cp基因组中分别存在23,931个密码子。有趣的是,亮氨酸(Leu)是PMA中最丰富的氨基酸,其次是异亮氨酸(Ile);而丝氨酸(Ser)是PIX中最丰富的氨基酸,其次是亮氨酸(Leu);两个物种中丰度最低的氨基酸是蛋氨酸。AUG是首选密码子,在PMA和PIX中编码蛋氨酸(Met)的RSCU分别为1.91和2.0,有31个(48.4%)和29个(45.3%)的密码子存在密码子偏向(RSCU > 1)。 图2:密码子偏好性分析 重复序列分析 PMA共检测到35个重复序列,其中正向重复19个、回文重复13个、反向重复3个,总共比PIX少5个。大多数重复序列分布在LSC区,其次是IRs区和SSC区。此外,位于蛋白质编码基因重复序列占多数,位于基因间间隔序列的重复序列占少数。PMA和PIX共发现61个和60个SSRs,检测到的SSRs包括单、二、三和四核苷酸重复序列,其中位于LSC区域的SSRs最多,其次是IR区域、SSC区域。此外,大多数SSRs存在于基因间间隔区,其次是PCGs和tRNA基因。 图3:PMA和PIX叶绿体基因组中重复序列和SSRs类型 比较基因组分析 将PMA和PIX叶绿体基因组的LSC/IR和SSC/IR与其他8个近缘种进行比较,结果发现rps19基因远离PPE和PPR的LSC/IRb边界或缺失,而rpl2基因跨越LSC/IRb边界,在PPE和PPR中分别向LSC区域延伸717 bp和737 bp。ycf1基因存在于所有cp基因组的IRa/SSC边界,ndhF基因存在于所有10个cp基因组的SSC区域。trnH基因在大多数物种中位于靠近IRa/LSC边界的LSC区域,而在PPE和PPR中缺失。此外,mVISTA分析结果显示PMA和PIX的cp基因组相对于其他8个cp基因组序列具有较高的一致性和基因顺序保守性。但10个Physalis叶绿体基因组之间仍存在一定的序列多样性。 图4:IR区收缩与扩张分析和mVISTA分析 在10个Physalis物种中,Pi值从0到0.024不等,平均值为0.00235。此次共检测到9个高度分化区域,核苷酸多样性(Pi)值范围为0.00725 ~ 0.024。其中6个区域位于LSC区,2个区域位于SSC区,1个区域位于IRa区。Ka/Ks结果表明有20个基因的Ka/Ks值小于1,且PMA和PPR之间大部分基因的Ka/Ks值相对较高。 图5:Pi多样性分析以及选择压力分析 系统发育树分析显示,所有物种形成3个主要聚类,分别对应3个亚族。在ML树中,聚类I包括来自Iochrominae亚族6属20种,其中Iochroma属10种,Dunalia属5种,Eriolarynx属2种,Acnistus属、Saracha属和Vassobia属各1种。PMA,PIX、PAN、PPU、PMI、PPH、PPE、PCH、PPR和PAF分为聚类II。聚类III包括来自Withania亚族的Withania coagulans、Withania adpressa和Withania riebeckii 3种。结果表明,PMA、PIX和PPH三者亲缘关系较近,PAF与其他Physalis种亲缘关系较远。 图6:系统发育树分析 结论 在本研究中首次测序并发表了PMA和PIX的叶绿体基因组序列,并与另外8个Physalis物种的cp基因组进行了比较。结果发现PMA和PIX的cp基因组在基因组结构、基因含量和序列上与其他8种酸浆属相似;此外还发现了一些核苷酸分化的热点可作为后续Physalis物种鉴定和系统发育分析的潜在DNA标记;对Physaleae族的系统发育研究首次在cp基因组水平提供了新的见解。本研究不仅提供了两个新的酸浆属cp基因组,而且可以为酸浆属的物种鉴定、系统发育分析和资源保护提供重要的参考数据。