2024-04-25
经过近半年的研发、测试与验证,派森诺全新一代代谢组学数据库——PerSonalbio Next-Generation Metabolomics Database(简称PSNGM)现已正式上线启用。该代谢数据库采用创新型模式构建,不仅来源丰富、容量巨大,更重要的是包含了医学、动物、植物、微生物、肠道菌群及中药6大分类库,95%以上代谢物可实现RT假阳性过滤,该库的研发旨在为客户提供更为精准、可靠且高深度的代谢组学分析结果,助力代谢组学技术在各研究领域取得更为卓越的研究成果。
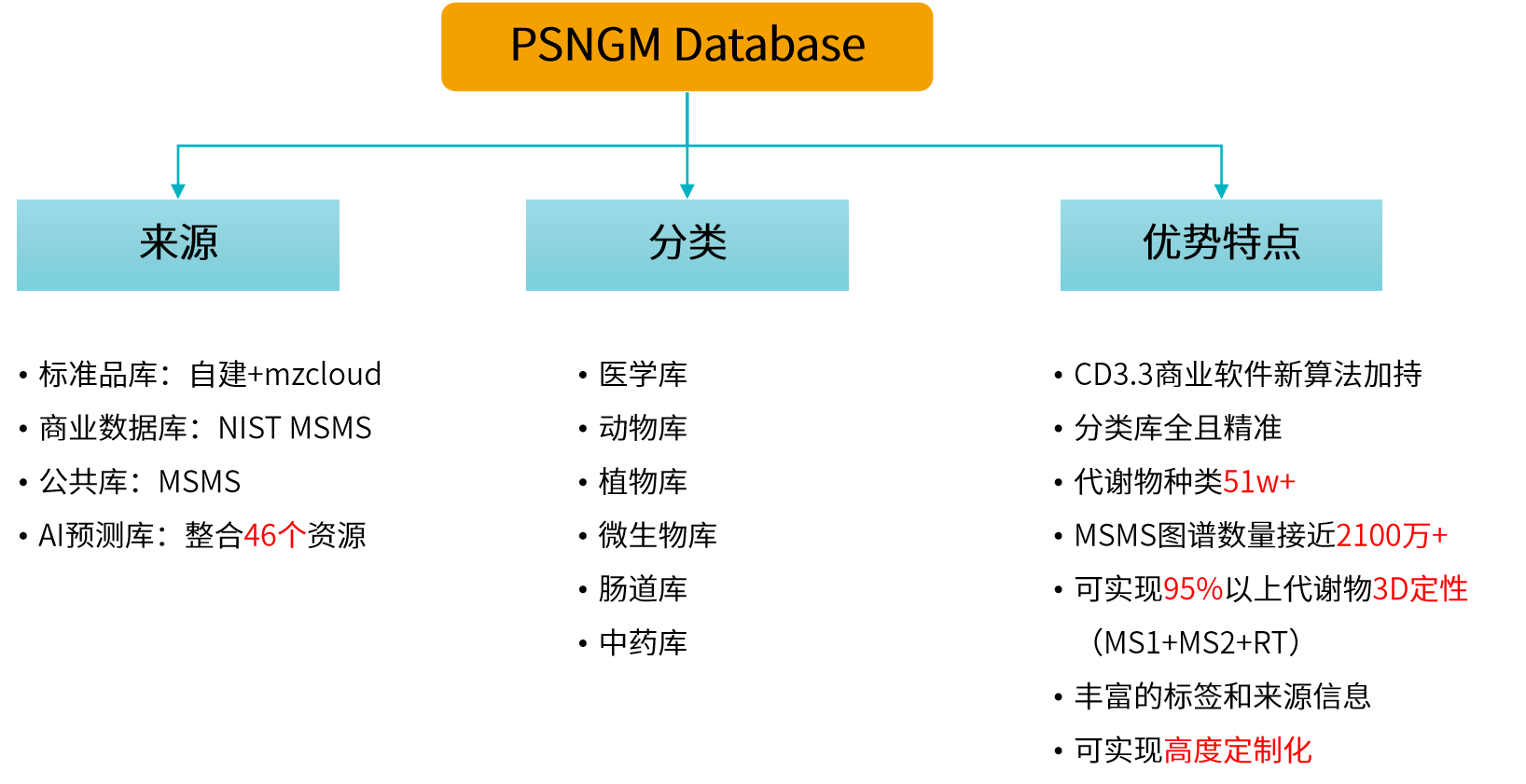
派森诺PSNGM代谢库组成及特点
PSNGM代谢数据库的主要特点
1、来源丰富,MSMS图谱数量庞大
除了传统的商业库、公共库及标准品自建库,PSNGM库集合了40+的资源库,利用精准、强悍的AI模型打造了庞大的MSMS图谱库,极大的补充了基于标准品库数量的限制,使得可靠的代谢物(Saleh Alseekh等[1]发布的最新鉴定标准:Level A-B)鉴定深度大幅提升。
2、分类齐全,涵盖各个研究领域
PSNGM库准确的将所包含的代谢物分为了6大类:医学、动物、植物、微生物、肠道菌群及中药库,几乎涵盖了生命科学各个研究领域,分库的关键作用可进一步提升代谢物鉴定的可靠性,降低假阳性几率。
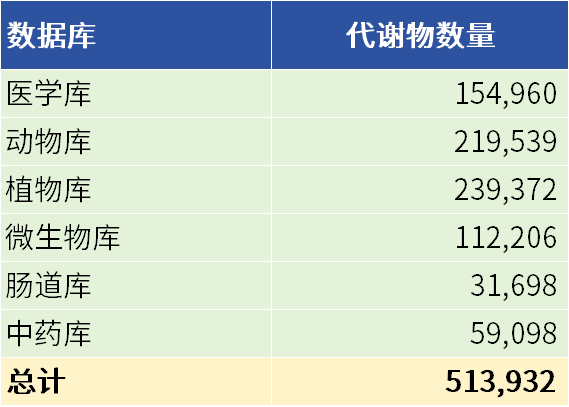
6大分类库容量
3、3D定性,让代谢物定性更准
PSNGM库采用先进AI算法,可实现库中95%以上代谢物具备可靠RT参考,可很好的降低代谢物鉴定的假阳性。LC-MS平台的一级质谱MS1、二级质谱MS2和保留时间RT是定性最关键的3个参数,绝大部分情况下只有自建标准品库才能实现3D定性,但自建标准品库由于标准品的原因容量非常有限。
Georgios Theodoridis[2]等人于2023年在国际知名期刊《Analytical Chemistry》上发表了一篇关于代谢组学的重要论文,文章集中揭露了基于LC-MS技术的代谢物鉴定中存在的行业乱象,列举了一系列典型错误案例,如将农药、杀虫剂等工业化学品误报为生物标志物,或将仅存在于植物体内的代谢物错误识别为哺乳动物的生物标记物等,这些现象在当前代谢组学研究中十分常见。代谢物定性过程中存在的准确性、高假阳性率等问题,亟待引起业界重视并寻求改善措施,而采用分类库和3D定性是直接有效的方法。
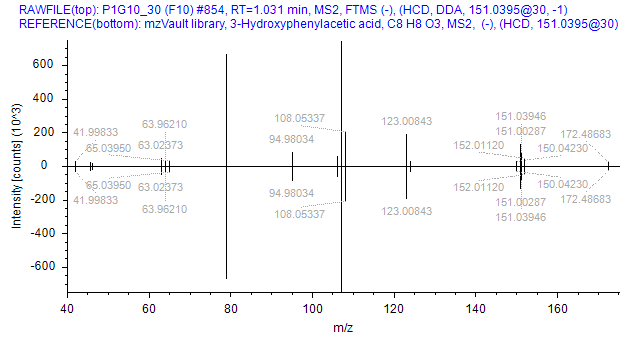
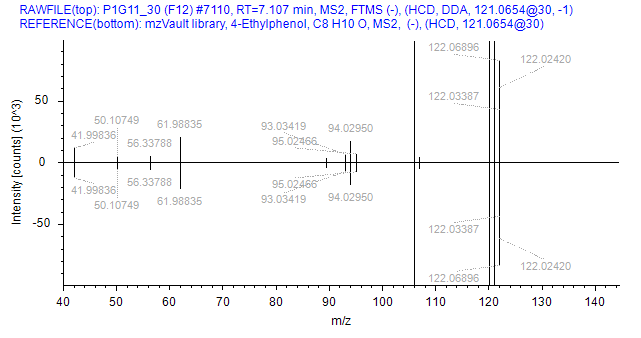

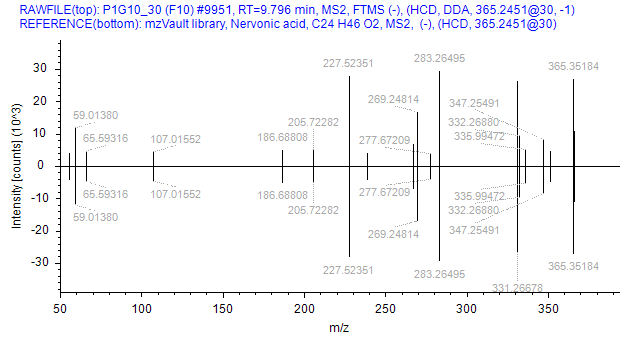
PSNGM库代谢物定性示例:镜像图上方为样品采集的实际图谱,下方为数据库中图谱
4、完善而丰富的标签信息,让结果解读更轻松
PSNGM库包含代谢物的化学分类、主流数据库ID、各类结构编码、Biogenic、DBSource、中文名等丰富的标签信息,让数据分析更便捷、更轻松。
5、个性化定制,让研究更精准
PSNGM代谢库的能力不仅仅局限于6大分类库,还可以实现个性化定制库,例如黄酮库、全氟和多氟烷基物质(PFASs)库、氧化脂质库、暴露组库、小分子药物库等,让研究结果更精准。
历经数十年发展,代谢组学已取得显著进步,但其深入应用仍受制于两大核心瓶颈:一是代谢物鉴定的深度不足,目前仅能触及约5-10%的鉴定率[3];二是鉴定的准确性受限,由于同分异构体等复杂情况导致假阳性结果频现。这两方面困境在很大程度上取决于所使用代谢数据库的容量规模与功能性能。正因如此,派森诺公司推出了新一代数据库PSNGM,其设计宗旨在于针对性地突破上述瓶颈,显著提升代谢物鉴定的广度与精度。
现实研究中,部分研究者易于陷入片面追求代谢物数量而忽视鉴定准确性的思维陷阱。须知,若代谢物定性环节即存在偏差,更多的鉴定数量非但无法增益研究,反而可能导致研究结果偏离,乃至得出错误的结论与见解。这一问题已引起学术界的广泛关注,如Georgios Theodoridis[2]等人明确呼吁:强化代谢物鉴定的严谨性与标准化报告至关重要,此举将有力巩固代谢组学作为一门科学的内在价值与可信度。PSNGM代谢库现已正式上线启用,其仍在不断完善和优化中,期望能为更多学者的代谢组学研究提供更有利的保障和支持。
参考文献:
1.Alseekh S, Aharoni A, Brotman Y, et al. Mass spectrometry-based metabolomics: a guide for annotation, quantification and best reporting practices[J]. Nature methods, 2021, 18(7): 747-756.
2.Theodoridis G, Gika H, Raftery D, et al. Ensuring fact-based metabolite identification in liquid chromatography–mass spectrometry-based metabolomics[J]. Analytical Chemistry, 2023, 95(8): 3909-3916.
3.da Silva R R, Dorrestein P C, Quinn R A. Illuminating the dark matter in metabolomics[J]. Proceedings of the National Academy of Sciences, 2015, 112(41): 12549-12550.


