mBio 2015 IF:6.975
关键词:光学图谱技术;端粒;DNA 转座元件;第三代单分子测序技术
研究背景
真核生物中存在大量的重复序列,这些重复序列会对基因组组装造成很大的影响。传统的高通量测序技术受制于读长,无法很好地解析这些重复序列。第三代单分子测序技术平均读长可达到 10~15 kb,最长读长超过 60 kb,能很好地解析富含重复的区域。光学图谱技术可用于构建高分辨率的图谱。本研究采用第三代单分子测序技术结合光学图谱技术获得了一个 36 Mb 真菌接近完整的全基因组序列图谱。
实验方法
供试样本:棉花黄萎病菌 Verticillium dahliae;
文库大小:22,000 bp,采用 BluePippin 去除 7,000 bp 以下的片段;
测序数据: 14 SMRT Cell P4-C2 + 4 SMRT Cell P5-C3
测序平台:PacBio RS II;
研究结果
(2) 两步法拼接(第一步采用 A5MiSeq对 MiSeq 数据进行拼接;然后采用PBjelly利用 Pacbio 数据进行拼接)相比一步法(直接采用 SPAdes对MiSeq数据和Pacbio 数据进行拼接)拼接效果更好;
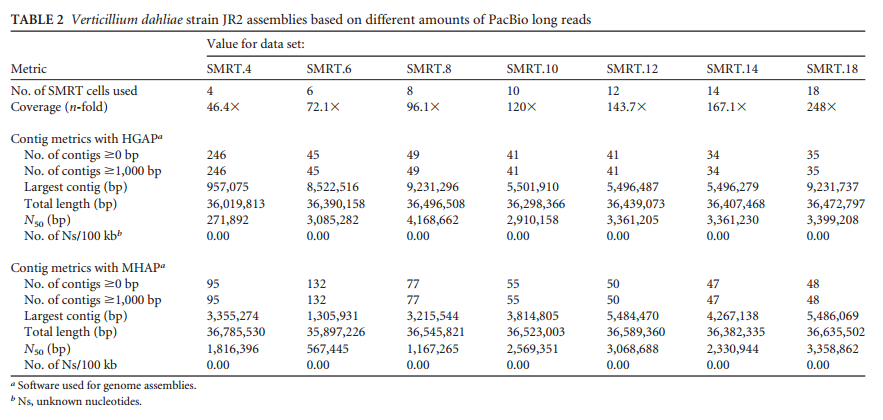
(3)Pacbio 测序数据对拼接效果(见表 1);
(4) 相比 MHAP 软件,HGAP 拼接效果更好(三代测序数据);
(5)当三代测序数据量超过 50 ×,数据量的增加对拼接结果;当测序数据量超过100 ×,数据量对拼接结果的改善效果不显著 (见表 2)。
参考文献
Luigi Faino , Michael F. Seidl , et al. Single-Molecule Real-Time Sequencing Combined with Optical Mapping Yields Completely Finished Fungal Genome. Mbio, 2014, 6 (4).
项目经验
拉丁名 | 基因组大小 | GC含量 | 派森诺已完成数目 |
Saccharomyces pastorianus | 22.3 | 39.25 | 1 |
Cerrena.sp | 43.66 | 47.48 | 1 |
Chaetomium thermophilum | 28.32 | 52.6 | 1 |
Aspergillus terreus | 29.36 | 52.2 | 1 |
colletotrichum orbiculare | 90.08 | 37.8 | 1 |
cladosporium sphaerospermum | 38.52 | 54.3 | 1 |
lentinula edodes | 41.36 | 46 | 1 |
Fusarium proliferatum | 44.18 | 48.4 | 2 |
表一
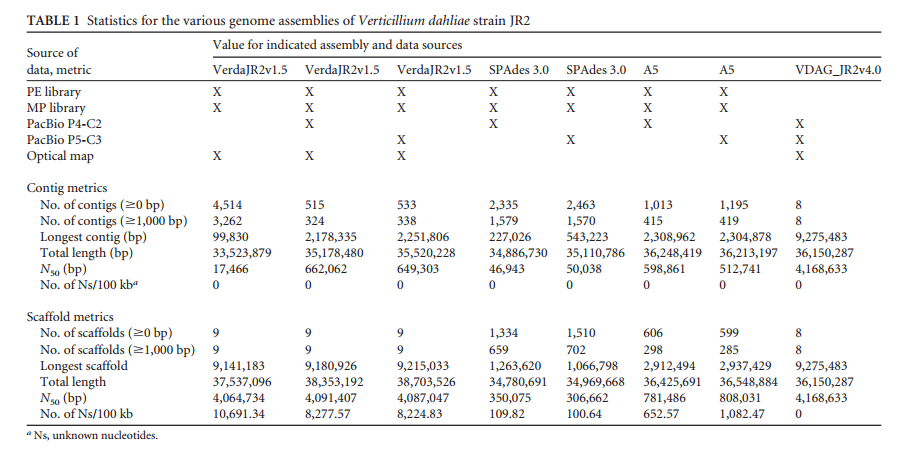
表二